
(c) 2017 ทิโมธี เบกเกอร์ และ วาน-ปิง ลี
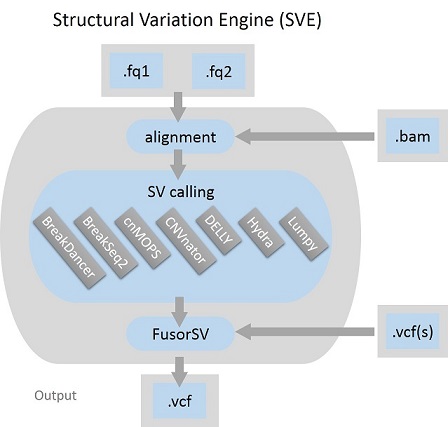
SVE คือกลไกการดำเนินการที่ใช้สคริปต์ Python สำหรับการตรวจจับ Structural Variation (SV) และสามารถใช้กับระดับอินพุตข้อมูล, FASTQ แบบดิบ, BAM ที่สอดคล้อง หรือรูปแบบการเรียกตัวแปร (VCF) และสร้าง VCF แบบรวมเป็นเอาต์พุต ตามการออกแบบ SVE ประกอบด้วยการจัดตำแหน่ง การปรับเปลี่ยน และชุดอัลกอริธึมการเรียก SV ที่ล้ำสมัยตามค่าเริ่มต้น ได้แก่ BreakDancer, BreakSeq, cnMOPS, CNVnator, DELLY, Hydra และ LUMPY นอกจากนี้ FusorSV ยังถูกฝังไว้ซึ่งเป็นแนวทางการขุดข้อมูลเพื่อประเมินประสิทธิภาพและรวมชุดการโทรจากชุดอัลกอริธึมการโทร SV
กรุณาตั้งค่าสภาพแวดล้อม ROOT
export ROOTSYS=/ROOT_Build_Path
export LD_LIBRARY_PATH=$LD_LIBRARY_PATH:$ROOTSYS/lib
git clone --recursive https://github.com/TheJacksonLaboratory/SVE.git
cd SVE
makeโปรดตรวจสอบไฟล์ส่วนหัวของ python2.7 และแก้ไข "CFLAGS_FUSOR_SV" ใน Makefile ไฟล์ส่วนหัวอาจอยู่ที่ "/usr/include/python2.7" และใช้ "CFLAGS_FUSOR_SV=-I /usr/include/python2.7" แทน
make FusorSV
หรือคุณสามารถติดตั้ง FusorSV ได้โดย setup.py
cd SVE/scripts/FusorSV/
python setup.py build_ext --inplace
tar -zxvf data.tar.gz
หรือจะใช้รูปภาพ Dockerfile และ Docker ก็ได้ โปรดทราบว่าอาจจำเป็นต้องใช้ sudo สำหรับการใช้งานนักเทียบท่า ทั้งนี้ขึ้นอยู่กับการตั้งค่าเครื่องของคุณ
cd SVE
docker build .
ดึงอิมเมจนักเทียบท่าจากที่เก็บ
docker pull wanpinglee/sve
SVE สร้างขึ้นบน /tools/SVE ตรวจสอบความช่วยเหลือได้ที่
/tools/SVE/bin/sve
การอ่านแบบสั้นใน FASTQ จะถูกแมปกับ FASTA ที่กำหนด และ BAM ที่เรียงลำดับจะถูกสร้างขึ้น
bin/sve align [options] -r <FASTA> <FASTQ1 [FASTQ2]>
หากการอ่านได้รับจากรูปแบบ BAM การจัดตำแหน่งใหม่จะทำการแมปการอ่านใหม่กับ FASTA และสร้าง BAM ที่เรียงลำดับ เราใช้ SpeedSeq เพื่อปรับเปลี่ยนใหม่ให้สำเร็จ
bin/sve realign -r <FASTA> <BAM>
มีอัลกอริธึมการโทร SV เจ็ดแบบที่สามารถใช้สำหรับการโทร SV VCF จะถูกสร้างขึ้น
bin/sve call -r <FASTA> -g <hg19|hg38|others> -a <breakdancer|breakseq|cnvnator|hydra|delly|lumpy|cnmops> <BAM [BAM ...]>
หลังจากการโทร แต่ละตัวอย่างอาจมี VCF หลายตัว ขึ้นอยู่กับจำนวนผู้โทรที่ใช้ โปรดรวบรวม VCF ของตัวอย่างในโฟลเดอร์
vcfs ควรใช้ SVE ID เพื่อระบุผู้โทร
| รหัส SVE | ผู้โทร |
|---|---|
| 4 | เบรกแดนเซอร์ (v1.4.5) |
| 9 | cn.MOPS (v1.20) |
| 10 | CNVnator (v0.3.3) |
| 11 | เดลลี่ (v2) |
| 14* | จีโนม STRiP |
| 17 | ไฮดรา |
| 18 | เป็นก้อน |
| 35 | เบรกซีค (v2.2) |
| 0 | ความจริง (ไม่จำเป็น) |
หมายเหตุ*: เนื่องจากปัญหาใบอนุญาต GenomeSTRiP จึงไม่ได้ฝังอยู่ใน SVE อย่างไรก็ตาม โมเดลเริ่มต้นของ FusorSV สามารถจัดการ GenomeSTRiP VCF ได้
ตัวอย่างไฟล์ vcf อินพุตสามารถจัดระเบียบได้ดังนี้ โปรดทราบว่า vcfFiles เป็นอาร์กิวเมนต์สำหรับ -i สำหรับ FusorSV
python scripts/FusorSV/FusorSV.py -f scripts/FusorSV/data/models/default.pickle -L DEFAULT -r <FASTA> -i <vcfFiles>/ -p <THREADS> -o <OUT_DIR>
ตาม S0.vcf โมเดลใหม่จะถูกสร้างขึ้นและ VCF จะถูกรวมเข้ากับโมเดลใหม่
python scripts/FusorSV/FusorSV.py -L DEFAULT -r <FASTA> -i <vcfFiles>/ -p <THREADS> -o <OUT_DIR>
โครงการนี้ได้รับอนุญาตภายใต้ใบอนุญาต GPL-3.0 โปรดดูใบอนุญาตสำหรับรายละเอียด