
โปรดทราบว่ารหัสของโพไซดอนเพิ่งถูกถ่ายโอนไปยัง NextFlow ดังนั้นอาจมีข้อบกพร่องบางอย่าง โปรดอย่าลังเลที่จะรายงานปัญหา!
ที่นี่เรานำเสนอ Poseidon ซึ่งเป็นไปป์ไลน์เพื่อตรวจจับไซต์ที่เลือกเป็นบวกอย่างมีนัยสำคัญและเหตุการณ์การรวมตัวกันใหม่ที่เป็นไปได้ในการจัดเรียงลำดับการเข้ารหัสโปรตีนหลายลำดับ ไซต์ที่ได้รับการคัดเลือกในเชิงบวกให้ข้อมูลเชิงลึกเกี่ยวกับประวัติวิวัฒนาการของลำดับของคุณเช่นการแสดงจุดร้อนการกลายพันธุ์ที่สำคัญสะสมเป็นผลมาจาก การแข่งขัน ของไวรัสโฮสต์ในระหว่างการวิวัฒนาการ
Poseidon อาศัยเครื่องมือของบุคคลที่สามที่แตกต่างกันหลากหลาย (ดูด้านล่าง) แต่ไม่ต้องกังวลเราห่อหุ้มเครื่องมือแต่ละตัวในคอนเทนเนอร์ Docker ของตัวเองและเชื่อมต่อในระบบการจัดการเวิร์กโฟลว์ NextFlow
ไปที่ตัวอย่างเล็ก ๆ ของการส่งออกโพไซดอนสำหรับโปรตีน spike SARS-COV-2 เมื่อเปรียบเทียบกับการศึกษาล่าสุดของ Zhou et al 2020.
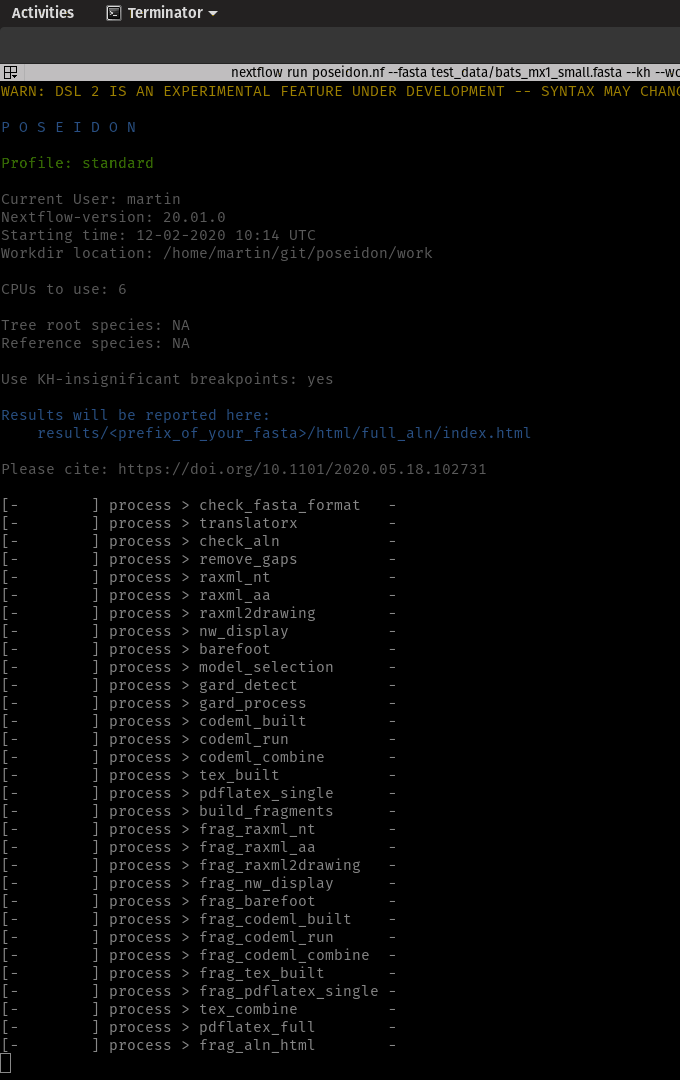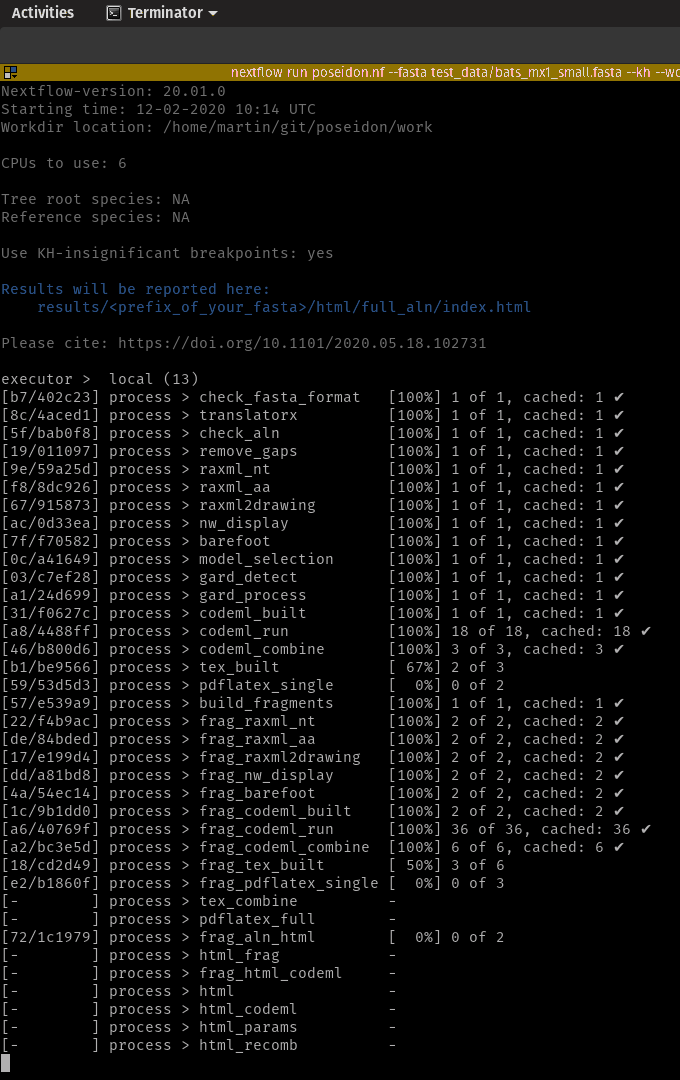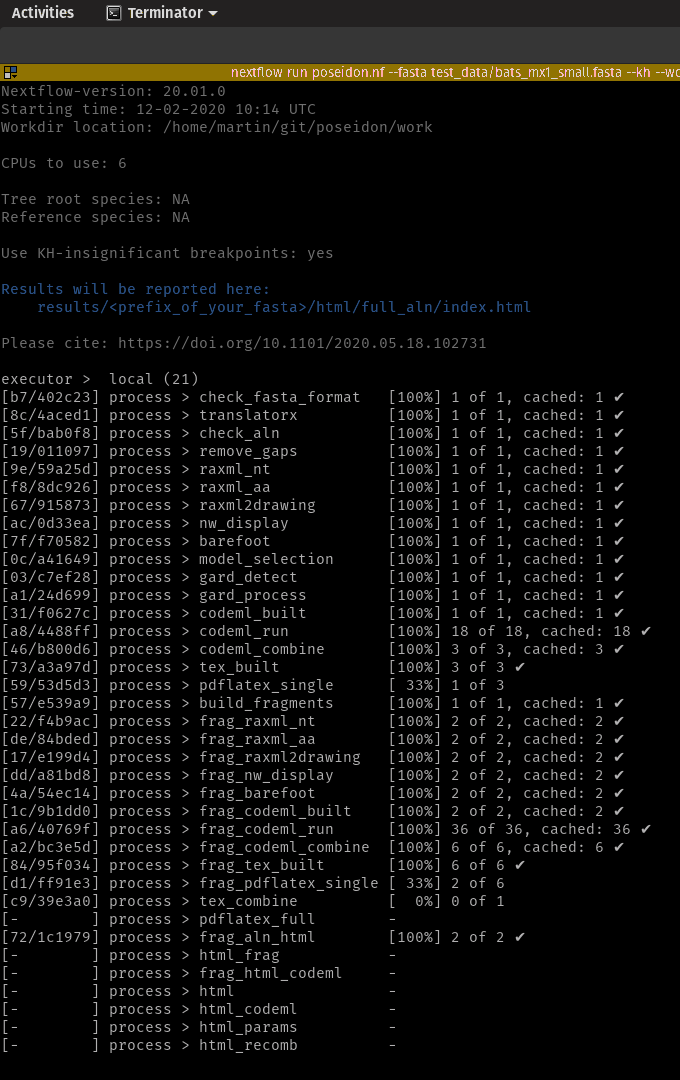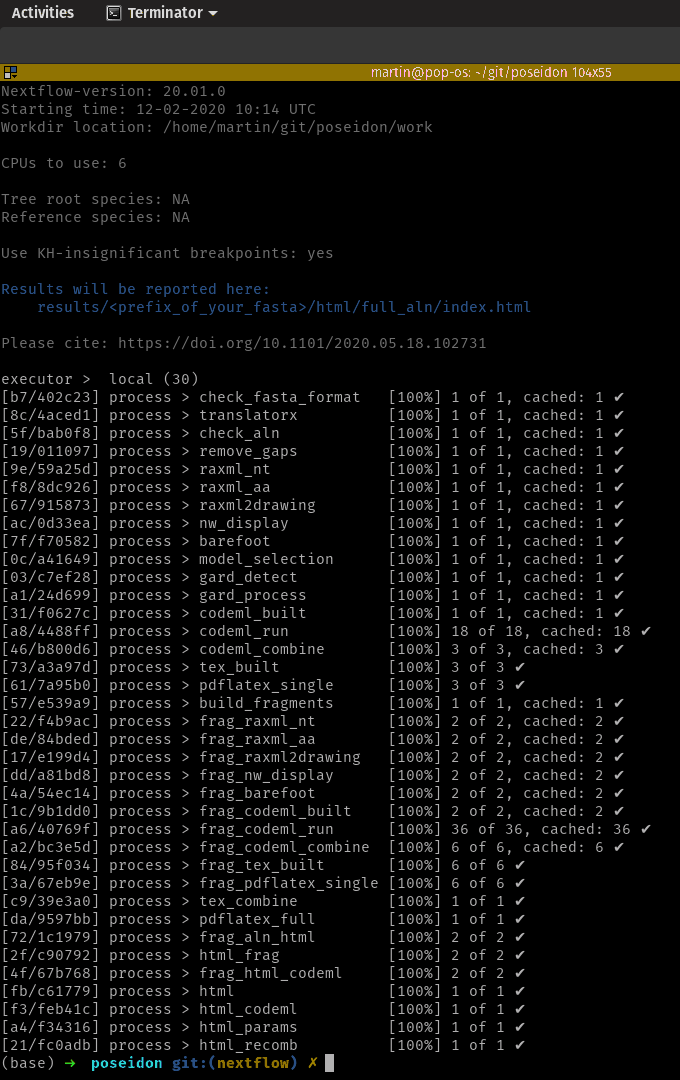
คุณต้องการเพียง NextFlow (เวอร์ชัน 20.+) และ Docker ที่ติดตั้งเพื่อเรียกใช้ Pipeline การพึ่งพาทั้งหมดจะถูกดึงโดยอัตโนมัติ
Run Poseidon โดยการโคลนที่เก็บนี้:
git clone https://github.com/hoelzer/poseidon.gitcd Poseidon NextFlow Run poseidon.nf -help
หรือปล่อยให้ NextFlow ทำการดึง
NextFlow Pull Hoelzer/Poseidon
เราขอแนะนำให้ใช้โพไซดอนเฉพาะ
#pullnextflow ดึง hoelzer/poseidon -r v1.0.1#runnextflow เรียกใช้ hoelzer/poseidon -r v1.0.1 -help
ขึ้นอยู่กับขั้นตอนการติดตั้งของคุณอัปเดตไปป์ไลน์ผ่าน git pull หรือ nextflow pull hoelzer/poseidon
สำคัญ: โพไซดอนต้องการลำดับนิวคลีโอไทด์ด้วยเฟรมการอ่านแบบเปิดที่ถูกต้องเป็นอินพุต นอกจากนี้ผลลัพธ์ขึ้นอยู่กับการเลือกลำดับของคุณอย่างมากดังนั้นคุณอาจพิจารณาเรียกใช้ท่อส่งหลายครั้งด้วยตัวอย่างที่แตกต่างกันของลำดับอินพุตของคุณ นอกจากนี้ท่อส่งไม่สามารถทำงานกับลำดับได้มากเกินไปเพราะในแกน Poseidon ใช้ codeml จากชุด PAML ที่ไม่ได้อยู่ในลำดับ> 100 ลำดับ โปรดค้นหาคำอธิบายโดยละเอียดของพารามิเตอร์อินพุตและการตั้งค่าด้านล่าง
NextFlow สามารถดำเนินการได้อย่างง่ายดายในสภาพแวดล้อมที่แตกต่างกันเช่นเครื่องในท้องถิ่นของคุณคลัสเตอร์ประสิทธิภาพสูงหรือคลาวด์ -profile ที่แตกต่างกันใช้เพื่อบอก nextflow ระบบควรใช้ระบบใด สำหรับการดำเนินการในท้องถิ่น -profile local,docker (และเป็นค่าเริ่มต้น) นอกจากนี้คุณยังสามารถเรียกใช้ Poseidon บน HPC โดยใช้ความเป็นเอกเทศผ่าน -profile lsf,singularity , -profile slurm,singularity หรือ -profile sge,singularity ในกรณีเช่นนี้โปรดพิจารณาที่จะปรับ --cachedir เพื่อชี้ไปที่ที่จะเก็บภาพเอกพจน์ในคลัสเตอร์ของคุณ พารามิเตอร์ --workdir อาจเป็นประโยชน์ในการปรับตำแหน่งที่จะจัดเก็บไดเรกทอรีการทำงานชั่วคราว (เช่นการใช้ /scratch แทน /tmp ขึ้นอยู่กับการกำหนดค่า HPC ของคุณ)
ตอนนี้สมมติว่าคุณใช้ NextFlow เพื่อดึงรหัส Poseidon และคุณเรียกใช้งานไปป์ไลน์บนเครื่องท้องถิ่นโดยใช้โปรไฟล์เริ่มต้น -profile local,docker
# show help nextflow เรียกใช้ hoelzer/poseidon -help # รันตัวอย่างเล็ก ๆ บนเครื่องท้องถิ่นด้วย # (ครั้งแรกนี้จะต้องใช้เวลามากขึ้นเนื่องจากดาวน์โหลดคอนเทนเนอร์ Docker) NextFlow Run Hoelzer/Poseidon -R v1.0.1 -Fasta ~/.nextflow/Assets/hoelzer/poseidon/test_data/bats_mx1_small.fasta -CORES 4# กลับมาทำงาน RunNextFlow Run Hoelzer/Poseidon -r v1.0.1 - -fasta ~/.nextflow/Assets/Hoelzer/Poseidon/test_data/bats_mx1_small.fasta -CORES 4 -Resume# แทนที่จะใช้คอร์ที่มีอยู่ทั้งหมดใช้จำนวนสูงสุดใน Machinenextflow ในท้องถิ่น Run Hoelzer/Poseidon -r v1.0.1 - -fasta ~/.nextflow/Assets/Hoelzer/Poseidon/test_data/bats_mx1_small. --max_cores 8 --cores 4
เพื่อทำซ้ำผลลัพธ์การเลือกเชิงบวกที่รายงานใน Fuchs และคณะ (2017), วารสารไวรัสวิทยาดำเนินการ:
NextFlow Run hoelzer/poseidon -r v1.0.0 -fasta ~/.nextflow/ทรัพย์สิน/hoelzer/poseidon/test_data/bats_mx1.fasta --cores 4-KH-OUTGROUP "PTEROPUS_ALECTO, EIDOLON_HELVUM, ROUSETTUS_AEGYPTIACUS, HYPSIGNATUS_MONSTROSUS"-การอ้างอิง "myotis_daubentonii"
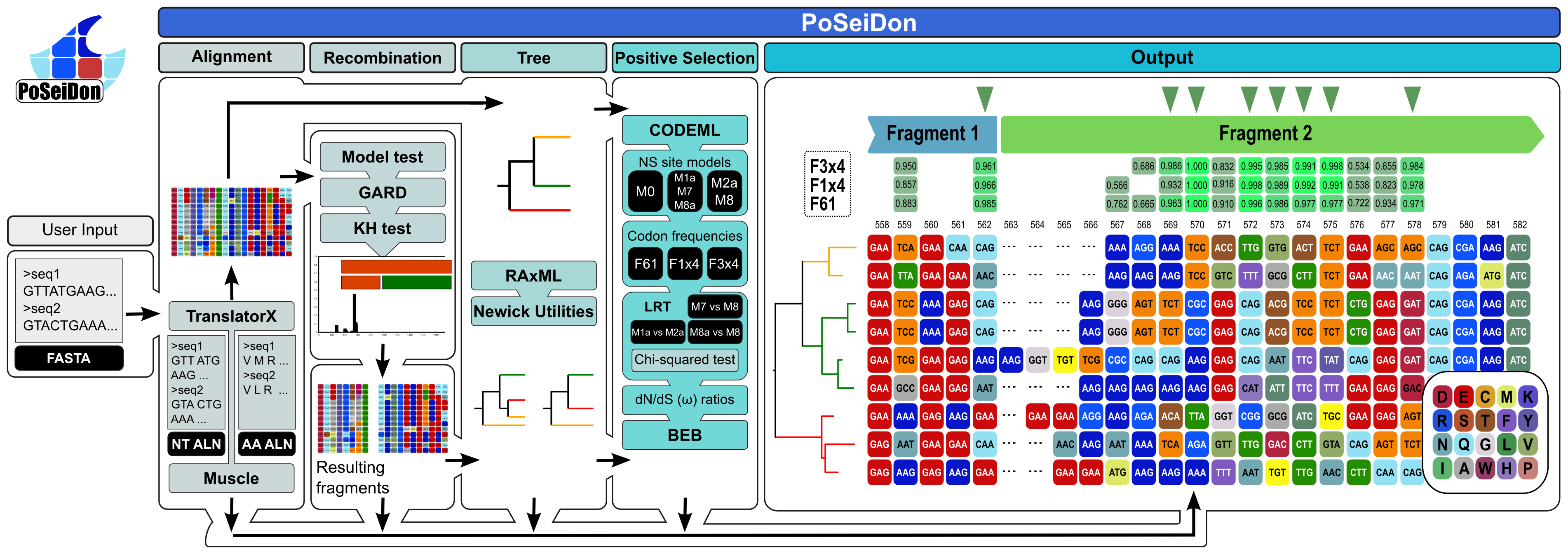
Poseidon Pipeline ประกอบด้วยการจัดตำแหน่งในเฟรมของลำดับการเข้ารหัสโปรตีนที่คล้ายคลึงกันการตรวจจับเหตุการณ์การรวมตัวกันใหม่และจุดพักวิวัฒนาการการสร้างสายวิวัฒนาการและการตรวจจับไซต์ที่เลือกในเชิงบวกในการจัดตำแหน่งเต็มรูปแบบและชิ้นส่วนที่เป็นไปได้ทั้งหมด ในที่สุดผลลัพธ์ทั้งหมดจะถูกรวมเข้าด้วยกันและมองเห็นได้ในหน้าเว็บ HTML ที่ใช้งานง่ายและชัดเจน ชิ้นส่วนการจัดตำแหน่งที่เกิดขึ้นจะถูกระบุด้วยแถบสีในเอาต์พุต HTML
Translatorx (v1.1), Abascal และคณะ (2010); 20435676
กล้ามเนื้อ (v3.8.31), เอ็ดการ์ (2004); 15034147
RAXML (v8.0.25), Stamatakis (2014); 24451623
Newick Utilities (v1.6), Junier และ Zdobnov (2010); 20472542
Modeltest, Posada และ Crandall (1998); 9918953
Hyphy (v2.2), Pond และคณะ (2005); 15509596
Gard, Pond และคณะ (2549); 17110367
paml/codeml (v4.8), หยาง (2007); 17483113
ทับทิม (v2.3.1)
Inkscape (v1.0)
pdftex (v3.14)
พารามิเตอร์ Poseidon ส่วนใหญ่เป็นทางเลือกและมีการอธิบายด้านล่างในรายละเอียด
--fasta
บังคับ. ไฟล์อินพุต fasta ของคุณจะต้องเป็นไปตามรูปแบบ:
>Myotis_lucifugus Mx1 Gene ATGGCGATCGAGATACGATACGTA... >Myotis_davidii Mx1 Gene ATGGCGGTCGAGATAAGATACGTT...
ลำดับทั้งหมดจะต้องมีเฟรมการอ่านแบบเปิดที่ถูกต้องได้รับอนุญาตให้มีอักขระนิวคลีโอไทด์เท่านั้น [A | C | G | T] และไม่มี codon หยุดภายใน
รหัสลำดับจะต้องไม่ซ้ำกันจนกว่าจะมีพื้นที่เกิดขึ้นครั้งแรก
--reference
ไม่จำเป็น. ค่าเริ่มต้น: ใช้ ID ลำดับแรกเป็นข้อมูลอ้างอิง คุณสามารถกำหนด ID สปีชีส์ หนึ่ง ไฟล์จากไฟล์ fasta หลายตัวของคุณเป็นสปีชีส์อ้างอิง ไซต์ที่เลือกในเชิงบวกและกรดอะมิโนที่สอดคล้องกันจะถูกวาดในส่วนที่เกี่ยวกับสายพันธุ์นี้ ID จะต้องตรงกับส่วนหัว Fasta จนกว่าจะเกิดขึ้นของพื้นที่แรก ตัวอย่างเช่นหากคุณต้องการ myotis lucifugus เป็นสปีชีส์อ้างอิงของคุณและไฟล์ fasta ของคุณมี:
>Myotis_lucifugus Mx1 Gene ATGGCGATCGAGATACGATACGTA...
ใช้
--reference "Myotis_lucifugus"
เป็นพารามิเตอร์ในการตั้งค่าสปีชีส์อ้างอิง ตามค่าเริ่มต้น ID แรกที่เกิดขึ้นในไฟล์ FASTA หลายตัวจะถูกใช้
--outgroup
ไม่จำเป็น. ค่าเริ่มต้น: ต้นไม้ไม่ได้รูท คุณสามารถกำหนด ID สปีชีส์สปีชีส์ หนึ่ง หรือ หลายตัว ) เป็นนอกกลุ่ม ต้นไม้สายวิวัฒนาการทั้งหมดจะถูกหยั่งรากตามสายพันธุ์นี้ ตัวอย่างเช่นหากไฟล์ fasta หลายตัวของคุณมี
ATGGCGATCGAGATACGATACGTA...
>Myotis_davidii Mx1 Gene
ATGGCGGTCGAGATAAGATACGTT...
>Pteropus_vampyrus Mx1 Gene
ATGGCCGTAGAGATTAGATACTTT...
>Eidolon_helvum Mx1 Gene
ATGCCCGTAGAGAATAGATACTTT...คุณสามารถกำหนด:
--outgroup "Pteropus_vampyrus,Eidolon_helvum"
เพื่อรูทต้นไม้ทั้งหมดที่เกี่ยวข้องกับสองสายพันธุ์นี้
--kh
ไม่จำเป็น. ค่าเริ่มต้น: เท็จ ด้วยพารามิเตอร์นี้คุณสามารถตัดสินใจได้ว่าควรคำนึงถึงจุดพักที่ไม่มีนัยสำคัญหรือไม่ จุดพักทั้งหมดได้รับการทดสอบสำหรับความไม่ลงรอยกันทอพอโลยีที่สำคัญโดยใช้การทดสอบ Kashino Hasegawa (KH) Kishino, H. และ Hasegawa, M. (1989) จุดพักที่ไม่มีนัยสำคัญของ KH มักเกิดขึ้นจากการเปลี่ยนแปลงของความยาวสาขาระหว่างเซ็กเมนต์ อย่างไรก็ตามการคำนึงถึงจุดพักที่มีนัยสำคัญของ KH อาจเป็นเรื่องที่น่าสนใจเพราะเราได้สังเกตเห็นเว็บไซต์ที่เลือกในเชิงบวกในชิ้นส่วนโดยไม่มีการแทรกซึมของทอพอโลยีที่สำคัญ ชิ้นส่วนที่มีนัยสำคัญ KH ถูกทำเครื่องหมายในผลลัพธ์สุดท้ายเนื่องจากอาจไม่เกิดขึ้นจากเหตุการณ์การรวมตัวกันใหม่ที่แท้จริง
ตามค่าเริ่มต้นเฉพาะจุดพักที่สำคัญที่ใช้สำหรับการคำนวณเพิ่มเติม
โปรดทราบว่าการใช้จุดพักที่ไม่มีนัยสำคัญสามารถขยายเวลาการทำงานของโพไซดอนจากนาทีถึงชั่วโมงขึ้นอยู่กับจำนวนจุดพักที่ตรวจพบ
โปรดดู --help สำหรับพารามิเตอร์อื่น ๆ (GARD, RAXML, ... ) และแจ้งให้เราทราบหากคุณต้องการการปรับแต่งเพิ่มเติม!
หาก Poseidon ช่วยคุณได้โปรดอ้างอิง:
Martin Hölzerและ Manja Marz, "Poseidon: ท่อส่งสัญญาณ Nextflow สำหรับการตรวจจับเหตุการณ์การรวมตัวกันใหม่และการเลือกเชิงบวก", OUP Bioinformatics (2020)