พื้นที่เก็บข้อมูลนี้มีรหัสและข้อมูลสำหรับการจัดหมวดหมู่ผิวหนังที่มีวิสัยทัศน์การมองเห็นด้วยการมองเห็นด้วย GPT-4V
อ่านกระดาษ
โปรดค้นหาคำตอบโดยละเอียดของแบบฟอร์ม GPT Model Data Mendeley
นม
ปิ
Daltonlens
JSON
Openai
ฐาน 64
แพนด้า
. ├── data # Contains subdirectories for processed data │ ├── all │ ├── all_resized │ ├── all_resized_brettel_protan_1 │ ├── all_resized_brettel_deutan_1 │ ├── all_resized_brettel_tritan_1 │ ├── bn │ ├── bn_resized │ ├── bn_resized_label │ ├── bn_resized_label_brettel_protan_1 │ ├── bn_resized_label_brettel_deutan_1 │ ├── bn_resized_label_brettel_tritan_1 │ ├── mm │ ├── mm_resized │ ├── mm_resized_label │ ├── mm_resized_label_brettel_protan_1 │ ├── mm_resized_label_brettel_deutan_1 │ ├── mm_resized_label_brettel_tritan_1 │ └── selected_images.xlsx # Image names ├── RAW # Contains raw data downloaded from ISCI Archive ├── result # Results from running API_calling will be stored here │ ├── 2_shot_brettel_protan_1 │ │ ├──rep 1 │ │ └── ... │ ├── 2_shot_brettel_deutan_1 │ └── ... ├── CVD_classification_GPT.py # Call OpenAI API for classification ├── CVD_convertor.py # Convert original image to CVD simulated image ├── data_labeling.py # Add label to the image for reference ├── data_resizing.py # Resize the data with the original aspect ratio ├── data_selection.py # Select data from RAW └── README.md
ภาพ dermoscopic ทั้งหมดจะถูกดาวน์โหลดจาก ISIC Archive
โครงการนี้มีวัตถุประสงค์เพื่อการวิจัยเชิงวิชาการเท่านั้น รหัสในที่เก็บนี้จะถูกปล่อยภายใต้ใบอนุญาต MIT หากคุณใช้ข้อมูลที่มีให้โปรดอ้างอิง ISIC Archive.s
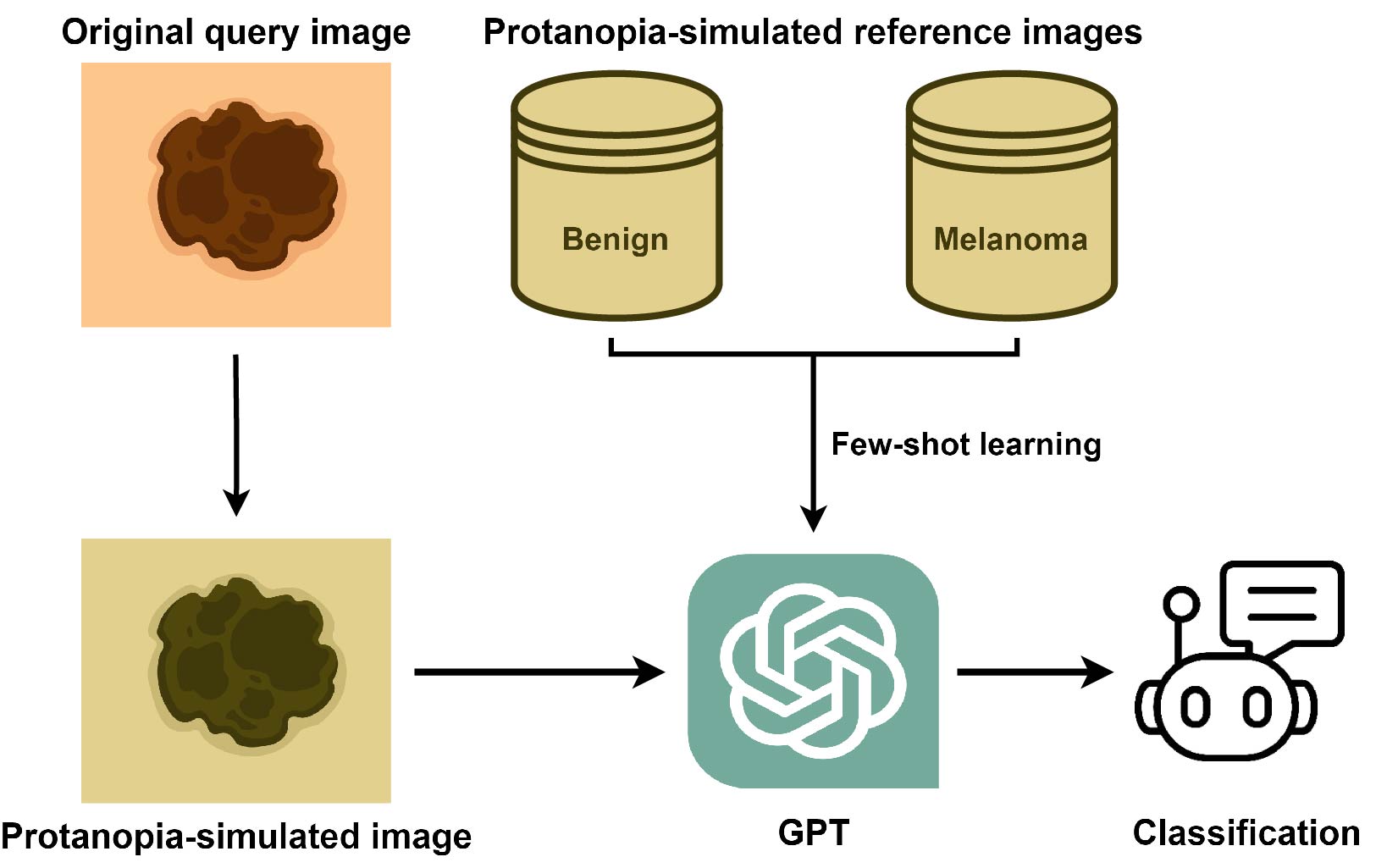
สำหรับการจำลอง CVD เราเลือกแพ็คเกจ Daltonlens-Python มันครอบคลุมอัลกอริทึมที่มีอยู่ในปัจจุบันของการจำลอง colorblindness บล็อกของผู้เขียนก็คุ้มค่าที่จะอ่าน
เราได้เลือก Brettel และคณะ 1997 เป็นวิธีการจำลอง CVD และตั้งค่าความรุนแรงที่ 1 ซึ่งสามารถปรับได้ด้วยรหัสที่ให้ไว้ตามต้องการ
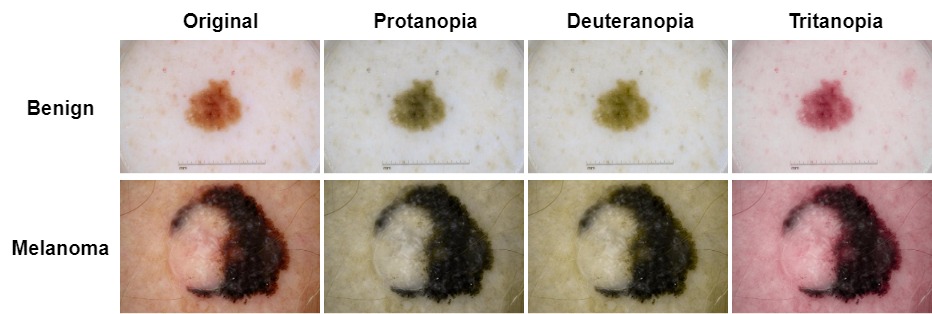
ตัวอย่างที่อ่อนโยน: ISIC_0012656
ตัวอย่าง Melanoma: ISIC_0046725
ดาวน์โหลดข้อมูลดิบจาก ISIC Archive
เลือกข้อมูล:
เรียกใช้ python data_selection.py -[ตัวเลือก]
ข้อมูลประมวลผล:
เรียกใช้ python data_resizeing.py -[ตัวเลือก]
เรียกใช้ python data_labeling.py -[ตัวเลือก]
แปลงข้อมูล:
เรียกใช้ python cvd_convertor.py -[ตัวเลือก]
โทร API:
เรียกใช้ Python cvd_classification_gpt.py -[ตัวเลือก]
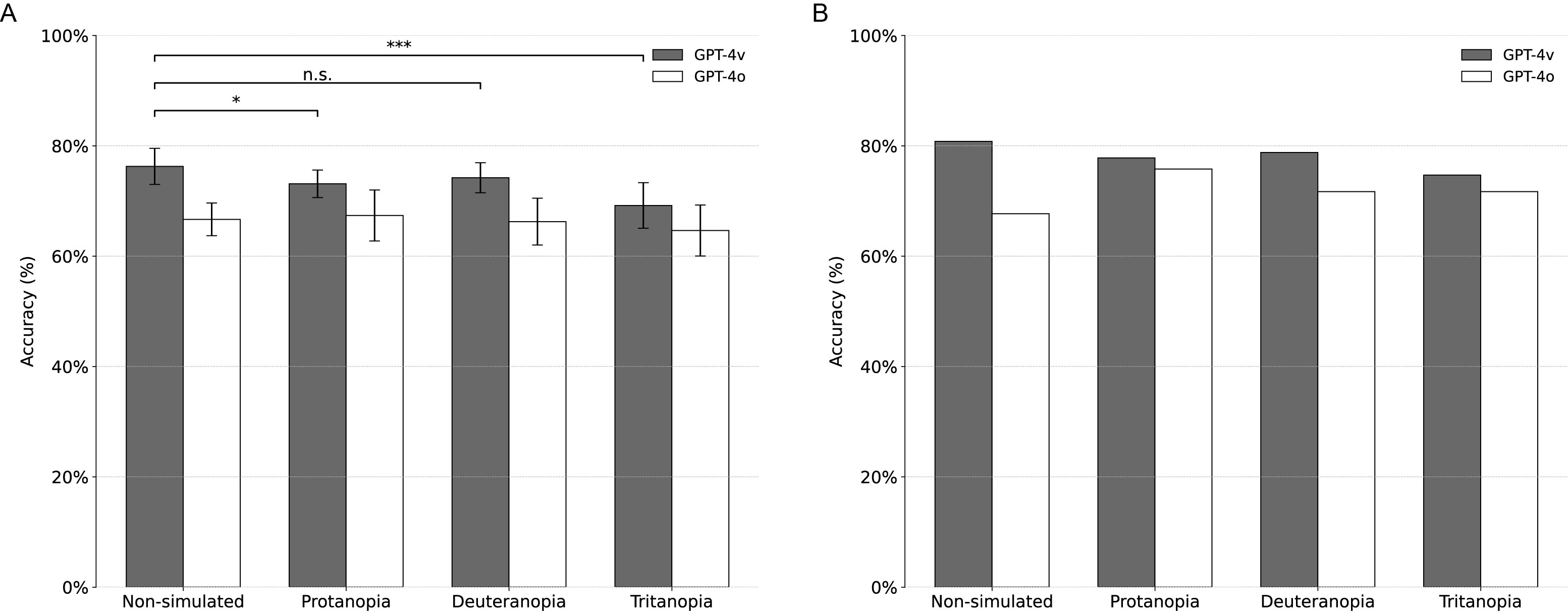
(a) ความถูกต้องการจำแนกประเภทเฉลี่ยของ GPT-4V สำหรับภาพที่ไม่ได้รับการจำลองและ CVD (Protanopia, Deuteranopia, Tritanopia) แถบข้อผิดพลาด: ส่วนเบี่ยงเบนมาตรฐาน *: P <0.05; **: P <0.01 (t-test; สองหาง) NS: ไม่สำคัญ การทดลองทั้งหมดมีการทำซ้ำสิบครั้ง (b) ความแม่นยำในการจำแนกประเภทของ GPT-4V หลังจากการประยุกต์ใช้กลยุทธ์ฉันทามติทั่วทั้งสิบซ้ำสำหรับการสืบค้นภาพแต่ละครั้ง GPT-4O รวมอยู่ในการเปรียบเทียบ
GPT-4V ปรับการตีความในการค้นพบสีตามการจำลอง CVD ที่แตกต่างกัน โดยเฉพาะอย่างยิ่งมันไม่ได้พูดถึงสีแดงหรือสีชมพูในการจำลอง Protanope และ Deuteranope แต่เชื่อมโยงกับสีเหล่านี้กับการทำนายมะเร็งผิวหนังในภาพที่ไม่ได้จำลอง ในการจำลอง Tritanopia-โดดเด่นด้วยเฉดสีสีชมพูที่แพร่หลาย-GPT-4V ไม่ได้รับการรักษาสีชมพูอีกต่อไปว่าเป็นเมลาโนมาอีกต่อไป
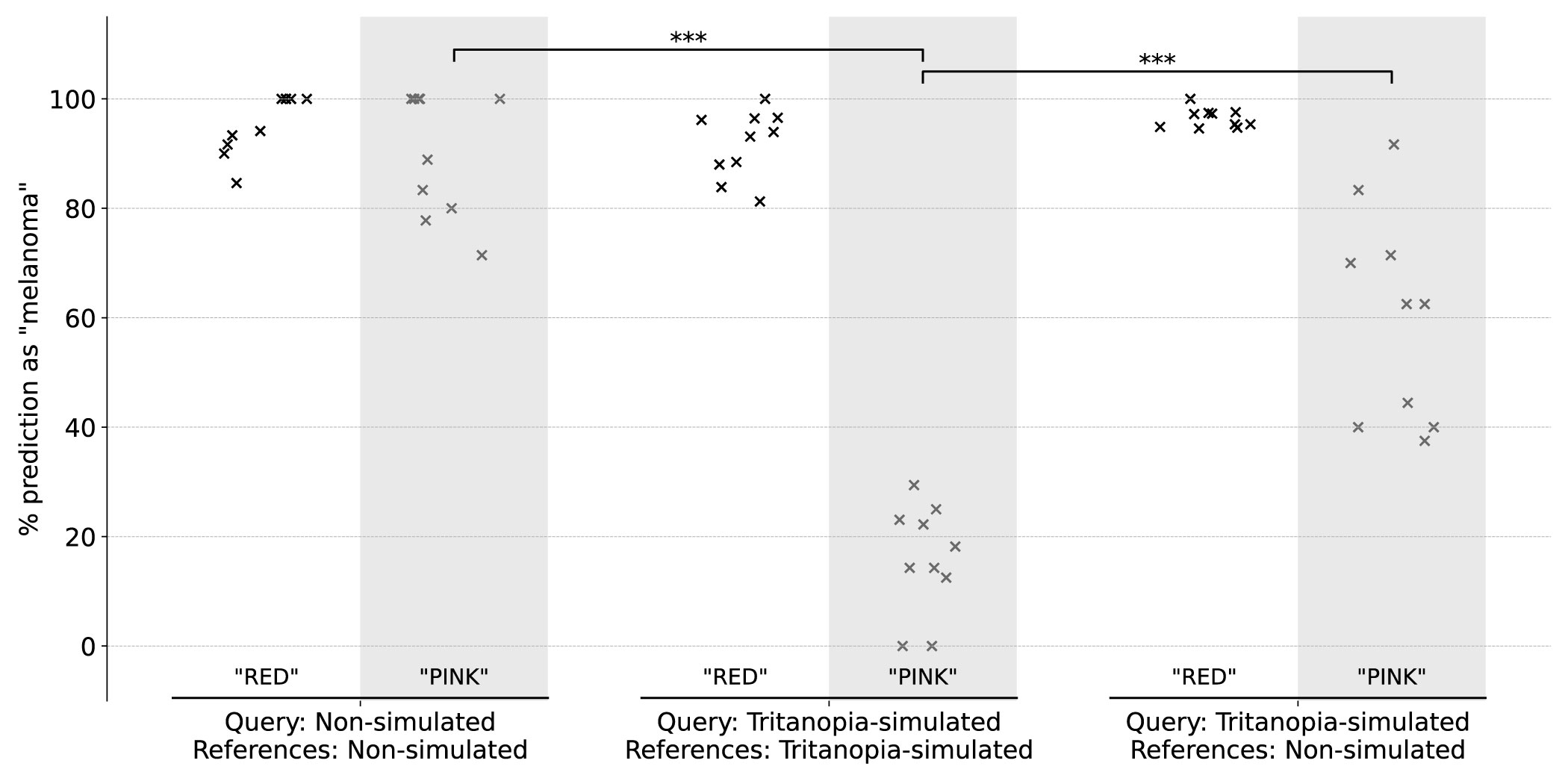
สำหรับเงื่อนไขที่ไม่มีการจำลองและการจำลองของ Tritanopia แต่ละครั้งจะมีการระบุการสอบถามภาพสองกลุ่มตามคำอธิบายของ GPT-4V สำหรับการคาดการณ์ กลุ่มแรกที่มีป้ายกำกับว่า“ สีแดง” รวมถึงคำอธิบายที่มีการกล่าวถึงสีแดงเพื่ออธิบายภาพการสืบค้นโดยไม่คำนึงถึงสีชมพู กลุ่มที่สองมีป้ายกำกับว่า“ สีชมพู” ประกอบด้วยคำอธิบายโดยเฉพาะการกล่าวถึงสีชมพูโดยไม่ต้องแดง แกน y แสดงเปอร์เซ็นต์ของการคาดการณ์“ melanoma” จากการทำซ้ำแต่ละครั้ง โดยค่าเริ่มต้นทั้งภาพการสืบค้นและการอ้างอิงนั้นไม่ได้จำลองหรือจำลองสถานการณ์ CVD เดียวกัน การทดสอบการระเหย (คอลัมน์สองคอลัมน์สุดท้าย) ทำเพื่อการจำลองการจำลองของ Tritanopia โดยการสุ่มตัวอย่างการอ้างอิงจากภาพที่ไม่ได้จำลอง ***: P <0.001 (t-test; สองหาง)