BigWig 檔案的快速批次資料加載,包含由 GPU 支援的表觀軌跡資料和相應序列,用於深度學習應用。
Bigwig-loader主要依賴rapidsai kvikio函式庫和cupy,這兩個函式庫最好使用conda/mamba安裝。 Bigwig-loader 現在也可以使用 conda/mamba 安裝。要建立安裝了 bigwig-loader 的新環境:
mamba create -n my-env -c rapidsai -c conda-forge -c bioconda -c dataloading bigwig-loader或將其新增至您的environment.yml 檔案:
name : my-env
channels :
- rapidsai
- conda-forge
- bioconda
- dataloading
dependencies :
- bigwig-loader並更新:
mamba env update -f environment.ymlBigwig-loader 也可以在已經安裝了rapidsai kvikio 函式庫和cupy 的環境中使用pip 安裝:
pip install bigwig-loader我們將 BigWigDataset 包裝在 PyTorch 可迭代資料集中,您可以直接使用:
# examples/pytorch_example.py
import pandas as pd
import torch
from torch . utils . data import DataLoader
from bigwig_loader import config
from bigwig_loader . pytorch import PytorchBigWigDataset
from bigwig_loader . download_example_data import download_example_data
# Download example data to play with
download_example_data ()
example_bigwigs_directory = config . bigwig_dir
reference_genome_file = config . reference_genome
train_regions = pd . DataFrame ({ "chrom" : [ "chr1" , "chr2" ], "start" : [ 0 , 0 ], "end" : [ 1000000 , 1000000 ]})
dataset = PytorchBigWigDataset (
regions_of_interest = train_regions ,
collection = example_bigwigs_directory ,
reference_genome_path = reference_genome_file ,
sequence_length = 1000 ,
center_bin_to_predict = 500 ,
window_size = 1 ,
batch_size = 32 ,
super_batch_size = 1024 ,
batches_per_epoch = 20 ,
maximum_unknown_bases_fraction = 0.1 ,
sequence_encoder = "onehot" ,
n_threads = 4 ,
return_batch_objects = True ,
)
# Don't use num_workers > 0 in DataLoader. The heavy
# lifting/parallelism is done on cuda streams on the GPU.
dataloader = DataLoader ( dataset , num_workers = 0 , batch_size = None )
class MyTerribleModel ( torch . nn . Module ):
def __init__ ( self ):
super (). __init__ ()
self . linear = torch . nn . Linear ( 4 , 2 )
def forward ( self , batch ):
return self . linear ( batch ). transpose ( 1 , 2 )
model = MyTerribleModel ()
optimizer = torch . optim . SGD ( model . parameters (), lr = 0.01 )
def poisson_loss ( pred , target ):
return ( pred - target * torch . log ( pred . clamp ( min = 1e-8 ))). mean ()
for batch in dataloader :
# batch.sequences.shape = n_batch (32), sequence_length (1000), onehot encoding (4)
pred = model ( batch . sequences )
# batch.values.shape = n_batch (32), n_tracks (2) center_bin_to_predict (500)
loss = poisson_loss ( pred [:, :, 250 : 750 ], batch . values )
print ( loss )
optimizer . zero_grad ()
loss . backward ()
optimizer . step ()可以從bigwig_loader.dataset匯入與框架無關的 Dataset 物件。該資料集物件傳回 cupy 張量。 Cupy 張量遵循 cuda 陣列接口,可以零複製轉換為 JAX 或張量流張量。
from bigwig_loader . dataset import BigWigDataset
dataset = BigWigDataset (
regions_of_interest = train_regions ,
collection = example_bigwigs_directory ,
reference_genome_path = reference_genome_file ,
sequence_length = 1000 ,
center_bin_to_predict = 500 ,
window_size = 1 ,
batch_size = 32 ,
super_batch_size = 1024 ,
batches_per_epoch = 20 ,
maximum_unknown_bases_fraction = 0.1 ,
sequence_encoder = "onehot" ,
)有關更多範例,請參閱範例目錄。
該庫用於加載具有相同維度的批量數據,這允許一些可以加快加載過程的假設。從下圖可以看出,當載入少量資料時,pyBigWig 速度非常快,但沒有利用資料載入的批次特性進行機器學習。
在下面的基準測試中,我們還使用 pyBigWig 建立了 PyTorch 資料載入器(使用 set_start_method('spawn')),以與每個 GPU 使用多個 CPU 的實際場景進行比較。我們看到 CPU 資料載入器的吞吐量並不隨 CPU 數量線性增加,因此很難獲得所需的吞吐量來保持 GPU 訓練神經網路在學習步驟期間飽和。
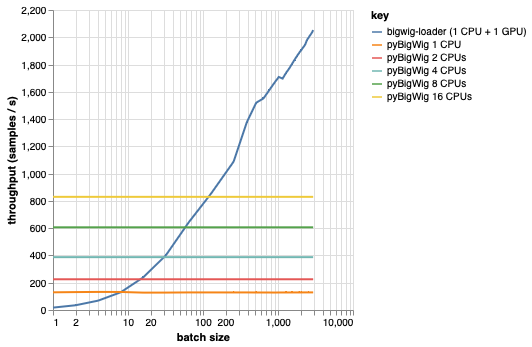
這就是bigwig-loader 解決的問題。這是如何使用 bigwig-loader 的範例:
git clone [email protected]:pfizer-opensource/bigwig-loadercd bigwig-loaderconda env create -f environment.yml在此環境中,您應該能夠運行pytest -v並看到測試成功。注意:您需要 GPU 才能使用 bigwig-loader!
本部分將引導您完成新增功能所需的步驟。如果有任何不清楚的地方,請提出問題。
git clone [email protected]:pfizer-opensource/bigwig-loadercd bigwig-loaderconda env create -f environment.ymlpip install -e '.[dev]'pre-commit install以安裝預提交掛鉤測試位於測試目錄中。最重要的測試之一是 test_against_pybigwig,它確保如果 pyBigWIg 中存在錯誤,那麼 bigwig-loader 中也會出現錯誤。
pytest -vv .當具有 GPU 的 github 運行程式可用時,我們也希望在 CI 中執行這些測試。但目前,您可以在本地運行它們。
如果您使用此庫,請考慮引用:
雷特爾、喬倫·塞巴斯蒂安、安德烈亞斯·珀爾曼、喬許·邱、安德烈亞斯·斯特芬和德約克·阿內·克萊維特。 “用於 BigWig 檔案表觀遺傳軌蹟的快速機器學習資料載入器。”生物資訊 40,沒有。 1(2024 年 1 月 1 日):btad767。 https://doi.org/10.1093/bioinformatics/btad767。
@article {
retel_fast_2024,
title = { A fast machine learning dataloader for epigenetic tracks from {BigWig} files } ,
volume = { 40 } ,
issn = { 1367-4811 } ,
url = { https://doi.org/10.1093/bioinformatics/btad767 } ,
doi = { 10.1093/bioinformatics/btad767 } ,
abstract = { We created bigwig-loader, a data-loader for epigenetic profiles from BigWig files that decompresses and processes information for multiple intervals from multiple BigWig files in parallel. This is an access pattern needed to create training batches for typical machine learning models on epigenetics data. Using a new codec, the decompression can be done on a graphical processing unit (GPU) making it fast enough to create the training batches during training, mitigating the need for saving preprocessed training examples to disk.The bigwig-loader installation instructions and source code can be accessed at https://github.com/pfizer-opensource/bigwig-loader } ,
number = { 1 } ,
urldate = { 2024-02-02 } ,
journal = { Bioinformatics } ,
author = { Retel, Joren Sebastian and Poehlmann, Andreas and Chiou, Josh and Steffen, Andreas and Clevert, Djork-Arné } ,
month = jan,
year = { 2024 } ,
pages = { btad767 } ,
}