該軟體包提供經過訓練的 U-net 模型用於肺部分割。目前有四種型號可供選擇:
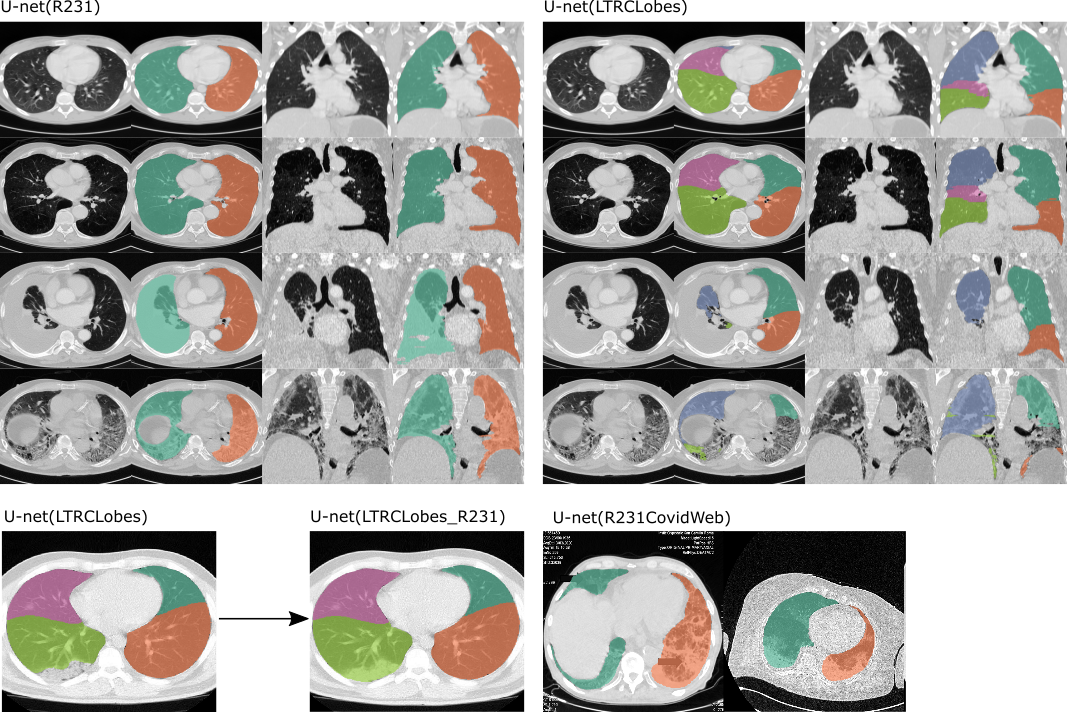
U-net(R231):該模型是在涵蓋廣泛視覺變數的大型且多樣化的資料集上進行訓練的。模型將各個切片分割,分別提取左右肺,包括氣囊、腫瘤和積液。氣管不會包含在肺分割中。 https://doi.org/10.1186/s41747-020-00173-2
U-net(LTRCLobes):該模型是在 LTRC 資料集的子集上進行訓練的。該模型對各個肺葉進行分割,但當存在密集的病變或每個切片上不可見裂隙時,其性能有限。
U-net(LTRCLobes_R231):這將運行 R231 和 LTRCLobes 模型並融合結果。 LTRCLobes 的假陰性將由 R231 預測填充並映射到鄰居標籤。 LTRCLobes 的誤報將會被刪除。融合過程需要大量計算,根據數據和結果,每個卷可能需要長達幾分鐘的時間。
U-net(R231CovidWeb)
兩種模型的應用實例。左: U-net(R231),將區分左肺和右肺,包括非常密集的區域,例如積液(第三行)、腫瘤或嚴重纖維化(第四行)。右: U-net(LTRLobes),將區分肺葉,但不包括非常密集的區域。 LTRCLobes_R231將融合 LTRCLobes 和 R231 結果。 R231CovidWeb使用額外的 COVID-19 資料進行訓練。
輸出的語意:
兩種標籤模型(左-右):
1 = 右肺
2 = 左肺
五個標籤模型(肺葉):
1 = 左上葉
2 = 左下葉
3 = 右上葉
4 = 右中葉
5 = 右下葉
有關肺部 CT 數據的更多令人興奮的研究,請查看我們研究小組的網站:https://www.cir.meduniwien.ac.at/research/lung/
如果您在工作中使用此程式碼或經過訓練的模型之一,請參閱:
Hofmanninger, J.、Prayer, F.、Pan, J. 等人。常規影像中的自動肺部分割主要是資料多樣性問題,而不是方法問題。歐洲無線電指數 4, 50 (2020)。 https://doi.org/10.1186/s41747-020-00173-2
本文包含對所使用資料集的詳細描述、對 U-net(R231) 模型的全面評估以及與參考方法的比較。
pip install lungmask
或者
pip install git+https://github.com/JoHof/lungmask
在 Windows 上,根據您的設置,可能需要提前安裝 torch:https://pytorch.org
僅 CPU 推理和 GPU 支援推理之間的運行時間差異很大。使用 GPU 處理一個磁碟區只需幾秒鐘,只使用 CPU 則需要幾分鐘。要使用 GPU,請確保您的手電筒安裝支援 CUDA。如果出現 cuda 記憶體不足錯誤,請使用可選參數--batchsize 1將批次大小減少到 1
lungmask INPUT OUTPUT
如果 INPUT 指向一個文件,則該文件將被處理。如果INPUT指向一個目錄,則會在該目錄中搜尋DICOM系列。找到的最大體積(就體素數量而言)將用於計算肺部掩模。 OUTPUT 是輸出檔名。支援所有 ITK 格式。
選擇型號:
預設使用 U-net(R231)。但是,您可以指定替代模型,例如 LTRCLobes...
lungmask INPUT OUTPUT --modelname LTRCLobes
其他選項,請輸入:
lungmask -h
from lungmask import LMInferer
import SimpleITK as sitk
inferer = LMInferer()
input_image = sitk.ReadImage(INPUT)
segmentation = inferer.apply(input_image) # default model is U-net(R231)
input_image 必須是 SimpleITK 物件。
載入替代模型,如下所示:
inferer = LMInferer(modelname="R231CovidWeb")
若要將模型融合功能用於(例如 LTRCLobes_R231),請使用:
inferer = LMInferer(modelname='LTRCLobes', fillmodel='R231')
從版本 0.2.9 開始,支援 numpy 陣列作為輸入磁碟區。此模式假設輸入 numpy 陣列的每個軸具有以下格式:
此模型僅適用於完整切片。要處理的切片必須顯示完整的肺部,並且肺部必須被組織包圍才能分割。然而,只要肺部被組織包圍,模型對於視野被裁剪的情況就相當穩定。
lungmask INPUT OUTPUT --modelname R231CovidWeb
常規 U-net(R231) 模型非常適合 COVID-19 CT 掃描。然而,來自網路的切片和病例報告的集合通常會被裁剪、註釋或以常規影像格式編碼,因此只能估計原始的亨斯菲爾德單位 (HU) 值。 U-net(R231CovidWeb) 模型的訓練資料透過從常規成像格式映射回 HU 的 COVID-19 切片進行了增強。資料由 MedSeg (http://medicalsegmentation.com/covid19/) 收集和準備。雖然常規 U-net(R231) 對這些影像顯示出非常好的結果,但在某些情況下,該模型可能會產生稍微改進的分割效果。請注意,使用網路圖像時,您必須將圖像映射回 HU。這篇文章描述如何做到這一點。或者,您可以設定--noHU標籤。 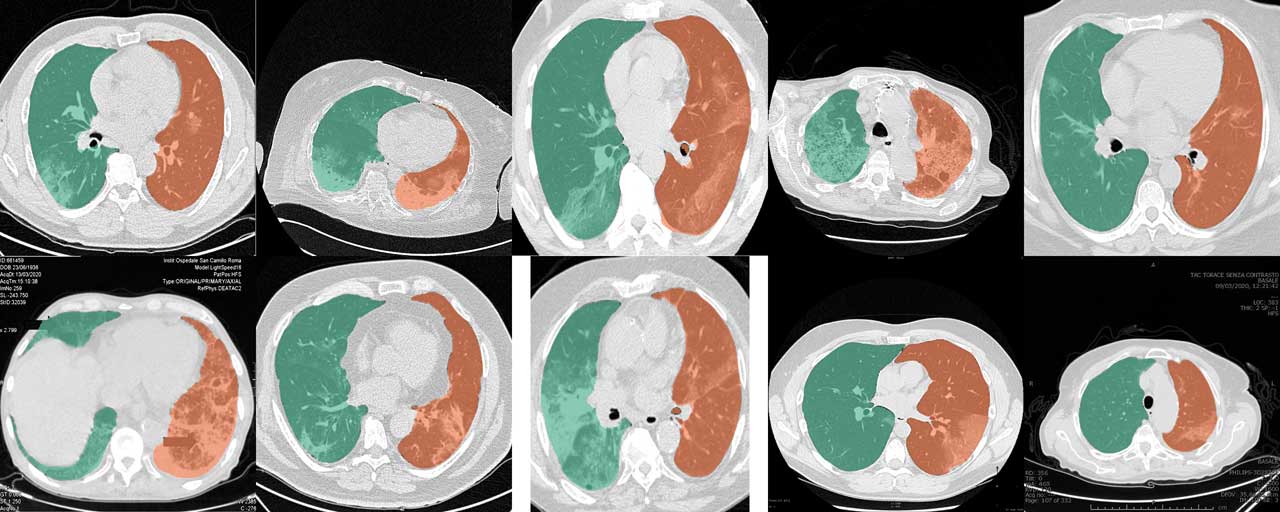
此功能僅在 0.2.5 和 0.2.14 之間的版本中可用。如果處理未以 HU 編碼的圖像,請使用--noHU標記。請記住,模型是根據 HU 中編碼的正確 CT 掃描進行訓練的。裁剪後的、帶有註釋的、非常高和非常低強度偏移影像的結果可能不是很可靠。使用--noHU標籤時,只能處理單一切片。