تم قياس تنفيذ DiffAbXL في الورقة: استكشاف درجات احتمالية السجل لتصنيف تصاميم تسلسل الأجسام المضادة.
| رتبة | نماذج | أبسي HER2 | طبيعة | الهدف من الألف إلى الياء-2 | افي.؟ | ||
|---|---|---|---|---|---|---|---|
| طلقة الصفر | التحكم في SPR | هيل | HER2 | ||||
| 1 | ديفأبكسل-A-DN | 0.43 | 0.22 | 0.62 | 0.37 | 0.41 | 0.41 |
| 2 | ديفأبكسل-A-SG | 0.46 | 0.22 | 0.64 | -0.38 | 0.43 | 0.274 |
| 3 | ديفأبكسل-H3-DN | 0.49 | 0 | 0.52 | -0.08 | 0.37 | 0.26 |
| 4 | IgBlend (الهيكل فقط) | 0.40 | 0.21 | 0.54 | -0.30 | 0.31 | 0.232 |
| 5 | مضاد الطي | 0.43 | 0.22 | 0.4 | -0.47 | 0.38 | 0.192 |
| 6 | ديفأبكسل-H3-SG | 0.48 | 0 | 0.4 | -0.41 | 0.29 | 0.152 |
| 7 | الإدارة السليمة بيئياً | 0.29 | 0 | 0 | 0.18 | 0.27 | 0.148 |
| 8 | ديفأب | 0.34 | 0.21 | 0 | -0.14 | 0.22 | 0.126 |
| 9 | أبلانج2 | 0.3 | 0 | 0 | -0.07 | 0.36 | 0.118 |
| 10 | IgBlend (التسلسل فقط) | 0.27 | 0 | 0 | -0.1 | 0.36 | 0.106 |
| 11 | ابلانج | 0.3 | 0 | 0 | -0.13 | 0.35 | 0.104 |
| 12 | dyMEAN | 0.37 | 0.15 | 0 | 0 | 0 | 0.104 |
| 13 | أبكس | 0.28 | 0.19 | 0 | 0 | 0 | 0.094 |
| 14 | أنتيبيرتي | 0.26 | 0 | 0 | -0.17 | 0.35 | 0.088 |
| 15 | يقصد | 0.36 | 0 | 0 | 0.02 | 0 | 0.076 |
| 16 | الإدارة السليمة بيئياً-IF | 0 | -0.27 | 0 | -0.53 | 0.42 | -0.076 |
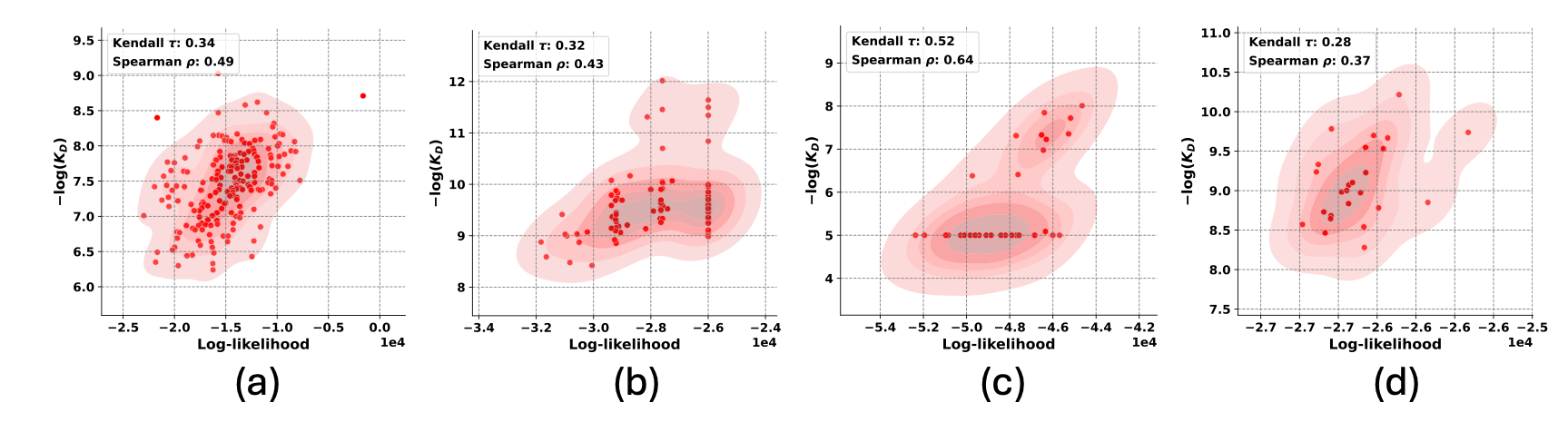
الشكل 1: نتائج DiffAbXL: أ) DiffAbXL-H3-DN لبيانات Absci ذات الطلقة الصفرية HER2 ب) DiffAbXL-A-SG لـ AZ Target-2، ج) DiffAbXL-A-SG لـ Nature HEL، د) DiffAbXL- A-DN للطبيعة HER2.
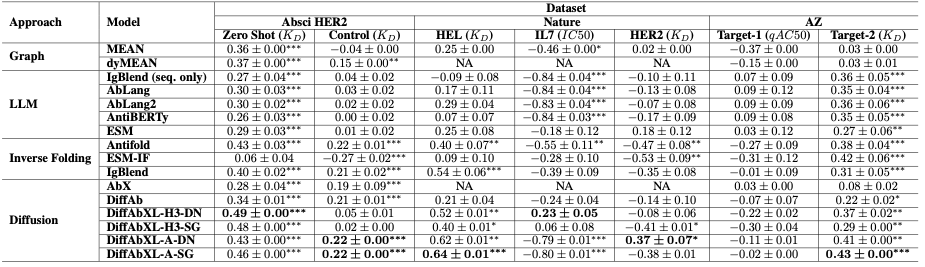
الجدول 1: ملخص نتائج ارتباط سبيرمان. الاختصارات: DN: وضع De Novo، SG: وضع توجيه الهيكل، NA: مطلوب Epitope أو بنية معقدة، ولكنها غير متوفرة. *، **، *** تشير إلى قيم p أقل من 0.05 و0.01 و1e-4 على التوالي.
لتسهيل علينا تقييم النموذج الخاص بك، نوصي بتنفيذ واجهة كطريقة Python في فصل يمكننا دمجه بسهولة مع مسار التقييم الخاص بنا. يجب أن تقبل الطريقة المدخلات التالية:
يجب أن تقوم الطريقة بإرجاع قاموس يحتوي على:
فيما يلي قالب أساسي في Python لتنفيذ هذه الواجهة:
def benchmark ( self , sequences , structure = None , mask = None , ** kwargs ):
"""
Benchmark the model on provided antibody sequences and structures.
Parameters:
sequences (list of str): List of antibody sequences.
structure (optional): Path to a PDB file. Currently, only one PDB file is provided per target dataset.
The PDB file may contain either just the antibody or an antibody-antigen complex,
depending on the dataset.
mask (optional): Binary list or array indicating the regions of interest in the sequences for metric calculations.
kwargs (optional): Additional parameters required by the model.
Returns:
dict: A dictionary containing log-likelihood scores and other relevant metrics.
"""
passيرجى التأكد من أن النموذج الخاص بك يُخرج درجات احتمالية السجل بتنسيق يمكننا استخدامه مباشرة لقياس تصميمات تسلسل الأجسام المضادة. سيساعدنا هذا في مقارنة أداء النموذج الخاص بك عبر مجموعات البيانات لدينا بكفاءة.
يوجد ملف تكوين واحد: sabdab.yaml، والذي يمكن استخدامه لتغيير أي من المعلمات. يمكنك تدريب النموذج باستخدام:
python train.py # For training.
- Train.py
- سرك
|-model.py
- التكوين
|-sabdab.yaml
- المرافق
|-load_data.py
|-arguments.py
|-model_utils.py
|-loss_functions.py
...
- بيانات
|-her2
...
يمكن استخدام الوزن والتحيزات لتتبع التجارب. يتم إيقاف تشغيله بشكل افتراضي، ولكن يمكن تشغيله عن طريق تغيير الخيار في ملف التكوين في ./config/sabdab.yaml
@article {Ucar2024.10.07.617023,
author = {Ucar, Talip and Malherbe, Cedric and Gonzalez Hernandez, Ferran},
title = {Exploring Log-Likelihood Scores for Ranking Antibody Sequence Designs},
elocation-id = {2024.10.07.617023},
year = {2024},
doi = {10.1101/2024.10.07.617023},
publisher = {Cold Spring Harbor Laboratory},
URL = {https://www.biorxiv.org/content/early/2024/10/24/2024.10.07.617023},
eprint = {https://www.biorxiv.org/content/early/2024/10/24/2024.10.07.617023.full.pdf},
journal = {bioRxiv}
}
إذا كنت تستخدم DiffAbXL في دراستك وعملك، فيرجى الاستشهاد به باستخدام ما يلي:
@Misc{talip_ucar_2024_DiffAbXL,
author = {Talip Ucar},
title = {Exploring Log-Likelihood Scores for Ranking Antibody Sequence Designs},
URL = {https://github.com/AstraZeneca/DiffAbXL},
month = {October},
year = {since 2024}
}