Sandwood ist eine Sprache, ein Compiler und eine Laufzeit für JVM-basierte probabilistische Modelle. Es soll das Schreiben von Modellen in einer Sprache ermöglichen, die Java-Entwicklern vertraut ist. Die resultierenden Modelle haben die Form von Java-Objekten und sind somit gut abstrahierte Komponenten eines umfassenden Systems.
Bei einem herkömmlichen Bayes'schen Modell muss der Benutzer das Modell entwerfen und dann Inferenzcode für jede Operation implementieren, die er am Modell ausführen möchte. Dies führt zu einer Reihe von Problemen:
Die Erstellung von Inferenzcode ist technisch anspruchsvoll und zeitaufwändig. Dieser Schritt bietet die Möglichkeit, dass subtile Fehler eingeführt werden.
Wenn das Modell geändert wird, muss der Inferenzcode aktualisiert werden. Dies ist zudem zeitaufwändig und technisch anspruchsvoll und führt zu folgenden Problemen:
Es wirkt abschreckend auf die Modifizierung von Modellen.
Es ist möglich, dass verschiedene Inferenzoperationen aus dem Takt geraten, sodass einige am alten Modell und andere am neuen Modell arbeiten.
Dies stellt eine weitere Möglichkeit für Fehler dar, in den Inferenzalgorithmus einzudringen, wenn Benutzer versuchen, subtile Anpassungen am vorhandenen Code vorzunehmen.
Die probabilistische Programmierung überwindet diese Probleme, indem sie die Beschreibung von Modellen entweder mithilfe einer API oder einer domänenspezifischen Sprache (DSL) ermöglicht, wie dies bei Sandwood der Fall ist. Das Sandwood DSL wird kompiliert, um Java-Klassen zu erzeugen, die das Modell darstellen und alle erforderlichen Inferenzoperationen implementieren. Dies hat eine Reihe von Vorteilen:
Sandwood besteht aus jeweils 3 Komponenten in ihrem entsprechenden Verzeichnis:
Jedes Stück ist von den vorhergehenden Stücken abhängig. Jedes Komponentenverzeichnis enthält eine Maven-POM-Datei zum Erstellen der Komponente. Für den Compiler und das Plugin müssen diese mit install aufgerufen werden, um sie für spätere Phasen verfügbar zu machen, z. B. mvn clean install . Die Beispiele sollten nur als mvn clean package erstellt werden.
Nach der Installation von Sandwood gibt es derzeit drei Möglichkeiten, ein Modell zu kompilieren:
Um Sandwood über die Befehlszeile zu verwenden, nachdem der Compiler und die Laufzeit erstellt wurden, finden Sie Befehlszeilenskripte mit ähnlicher Funktionalität wie javac in commandline/SandwoodC/bin . Um dies zu verwenden, fügt der Benutzer normalerweise das bin-Verzeichnis zum Pfad hinzu und ruft dann sandwoodc.sh HMM.sandwood auf, um das HMM-Modell zu kompilieren. sandwoodc.sh -h oder sandwoodc.bat -h führt dazu, dass eine Beschreibung der Verwendung und der verfügbaren Optionen ausgedruckt wird.
Die gesamte Funktionalität von SandwoodC kann erreicht werden, indem die Methode compile in org.sandwood.compilation.SandwoodC aufgerufen und ein Array mit den Argumenten übergeben wird, die an die Befehlszeile übergeben worden wären.
Das Maven-Plugin kann verwendet werden, um die Kompilierung von Sandwood-Dateien automatisch auszulösen, wenn das abhängige Projekt erstellt wird. Um das Plugin verwenden zu können, müssen Sie die Sandwood-Laufzeitumgebung als Abhängigkeit hinzufügen und das Plugin zum Build hinzufügen. Dies wird durch folgende Ergänzungen zur POM-Datei erreicht:
<dependencies>
<dependency>
<groupId>org.sandwood</groupId>
<artifactId>sandwood-runtime</artifactId>
<version>0.3.0</version>
</dependency>
</dependencies>
<build>
<plugins>
<plugin>
<groupId>org.sandwood</groupId>
<artifactId>sandwoodc-maven-plugin</artifactId>
<version>0.3-SNAPSHOT</version>
<executions>
<execution>
<configuration>
<partialInferenceWarning>true</partialInferenceWarning>
<sourceDirectory>${basedir}/src/main/java</sourceDirectory>
</configuration>
<goals>
<goal>sandwoodc</goal>
</goals>
</execution>
</executions>
</plugin>
</plugins>
</build>`
Die Einbindung des Elements <sourceDirectory>${basedir}/src/main/java</sourceDirectory> weist das Plugin an, in welchem Verzeichnis nach Modellen gesucht werden soll. Weitere nützliche Flags sind:
debug Diese Option wird verwendet, um Debugging-Informationen von SandwoodC zu erhalten. Wenn Sie diese Option auf true setzen, generiert Sandwood einen Trace seiner Aktionen. Der Standardwert ist false . Beachten Sie, dass dieses Flag zum Debuggen von Fehlern mit der Compilerkonfiguration/dem Compiler dient, nicht mit dem zu kompilierenden Modell. Fehler und Warnungen in den Sandwood-Modelldateien werden immer vom Compiler zurückgegeben.
partialInferenceWarning Diese Option wird verwendet, um zu verhindern, dass SandwoodC fehlschlägt, wenn einige Inferenzschritte nicht erstellt werden können. Wenn Sie diese Option auf true setzen, generiert Sandwood nur Warnungen bei fehlenden Schritten. Der Standardwert ist false .
sourceDirectory Dieser Parameter legt fest, in welchem Verzeichnis nach Modelldateien gesucht werden soll. Innerhalb dieses Verzeichnisses können sich die Modelle in verschiedenen Paketen befinden.
outputDirectory Dieser Parameter legt fest, in welchem Verzeichnis der Java-Quellcode für die Modelle abgelegt werden soll. Der Standardwert ist ${project.build.directory}/generated-sources/sandwood .
calculateIndividualProbabilities Dieser Parameter gibt an, ob die Wahrscheinlichkeiten für jede in einer Schleife erstellte Zufallsvariable anstelle eines einzelnen Werts für alle Instanzen berechnet werden sollen. Der Standardwert ist false .
javadoc Dieser Parameter weist den Compiler an, JavaDoc zu generieren, um das Modell zu ergänzen. Der Standardwert ist false .
javadocDirectory Dieser Parameter gibt den Speicherort an, an dem die generierten Dateien platziert werden sollen.
executable Mit diesem Parameter kann eine alternative JVM angegeben werden, mit der der Sandwood-Compiler ausgeführt wird.
Im Folgenden finden Sie eine Einführung in das Schreiben von Sandwood-Modellen und in die Verwendung der resultierenden Klassen, die die Modelle implementieren.
Eine Übersicht über die Schritte, die ein Modell durchläuft, ist in diesem Diagramm zu sehen. Modelle beginnen als .sandwood Datei, die zu einer Reihe von Klassendateien kompiliert wird. Diese können mehrfach instanziiert werden, um mehrere Instanzen des Modells mit unterschiedlichen Konfigurationen zu generieren.
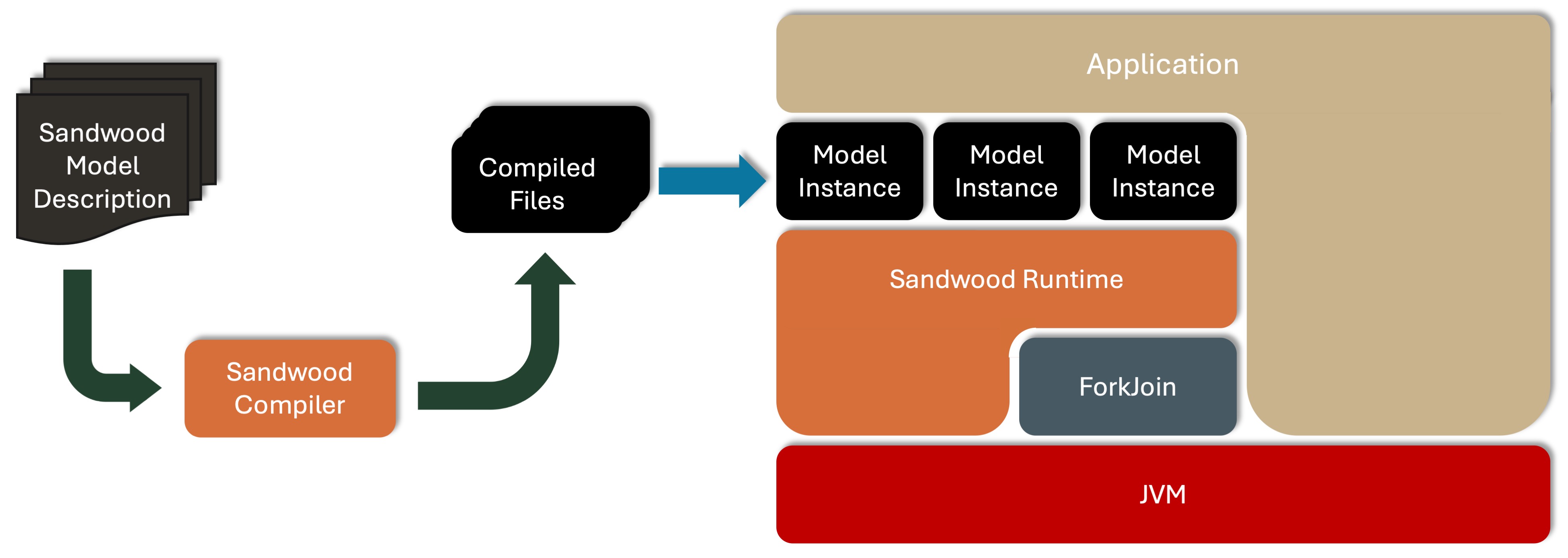
Als laufendes Beispiel verwenden wir ein Hidden-Markov-Modell (HMM). Dieses Modell ist hier in Sandwood geschrieben. Dieses Modell sollte in einer Datei namens HMM.sandwood im Paketverzeichnis org/sandwood/examples/hmm gespeichert werden. Eine ausführlichere Beschreibung der Sprache finden Sie hier.
package org . sandwood . examples . hmm ;
model HMM ( int [] eventsMeasured , int numStates , int numEvents ) {
//Construct a transition matrix m.
double [] v = new double [ numStates ] <~ 0.1 ;
double [][] m = dirichlet ( v ). sample ( numStates );
//Construct weighting for which state to start in.
double [] initialState = new Dirichlet ( v ). sample ();
//Construct weighting for each event in each state.
double [] w = new double [ numEvents ] <~ 0.1 ;
double [][] bias = dirichlet ( w ). sample ( numStates );
//Allocate space to record the sequence of states.
int sequenceLength = eventsMeasured . length ;
int [] st = new int [ sequenceLength ];
//Calculate the movements between states.
st [ 0 ] = categorical ( initialState ). sampleDistribution ();
for ( int i : [ 1. . sequenceLength ) )
st [ i ] = categorical ( m [ st [ i - 1 ]]). sampleDistribution ();
//Emit the events for each state.
int [] events = new int [ sequenceLength ];
for ( int j = 0 ; j < sequenceLength ; j ++)
events [ j ] = new Categorical ( bias [ st [ j ]]). sample ();
//Assert that the events match the eventsMeasured data.
events . observe ( eventsMeasured );
}Zusätzlich zur Dokumentation der Sandwood-Sprache und dem JavaDoc-Kommentar, der für ein Modell generiert werden kann, gibt es im Sandwood-Beispielverzeichnis eine Reihe von Beispielen. Wir empfehlen neuen Benutzern, diese zunächst zu untersuchen und zu ändern.
Eine Beschreibung der Sprache, die zur Beschreibung von Sandwood-Modellen verwendet wird, finden Sie hier. Die Sprache wurde mit der Absicht erstellt, Java-Entwicklern vertraut zu sein, verfügt jedoch nicht über die Möglichkeit, Objekte zu konstruieren. Wir planen, in Zukunft Unterstützung für Datensatztypen hinzuzufügen, um den Import und Export von Daten in und aus Modellen zu vereinfachen.
Wenn ein Modell kompiliert wird, werden eine Reihe von Klassendateien in demselben Paket generiert, in dem das Modell definiert ist. Eine dieser Klassen hat denselben Namen wie der dem Modell bereitgestellte Name, also in diesem Fall HMM.class und this ist die Klasse, die der Benutzer instanziieren sollte, um eine Instanz des Modells zu erhalten. Jede öffentlich sichtbare Variable im Modell entspricht einem Feld in der generierten Klasse. Das Beispiel-HMM ist unten zu sehen.
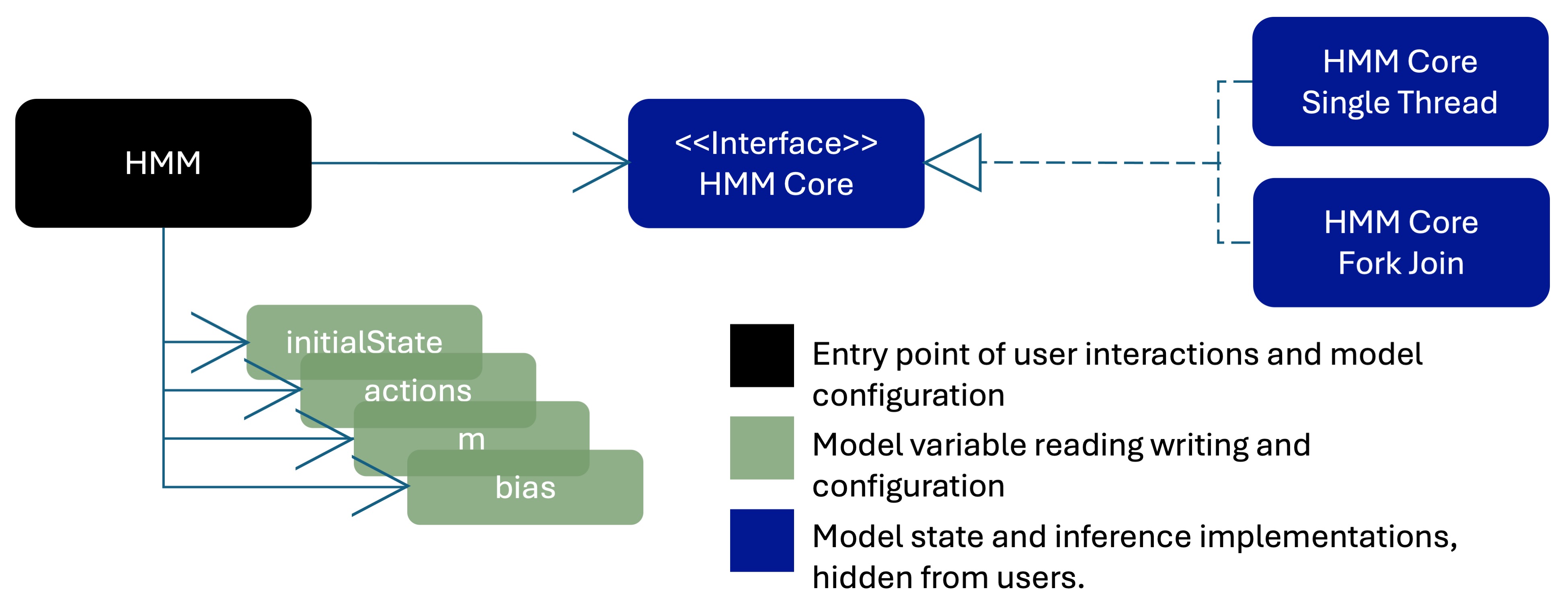
Durch Ausführen des Compilers mit gesetztem javadoc -Flag wird JavaDoc für jede öffentliche Methode und Klasse in der generierten Modelldatei erstellt.
Sobald das Modell kompiliert wurde, müssen wir Instanzen davon instanziieren. Diese Instanzen sind unabhängig und der Benutzer kann beliebig viele verschiedene Kopien des Modells erstellen.
Instanzen des Modellobjekts werden über den Klassenkonstruktor erstellt. Wie bereits beschrieben gibt es typischerweise drei Konstruktoren für das Modell. Der einzige Fall, in dem es weniger gibt, ist, wenn die verschiedenen Varianten des Konstruktors derselben Signatur zugeordnet sind. In diesem Fall gilt ein Konstruktor für mehr als eines dieser Szenarios.
Vollständiger Konstruktor – Dieser Konstruktor übernimmt alle Argumente, die in der Modellsignatur erscheinen, und legt sie fest. Dieser Konstruktor wird für die Operationen „Werte ableiten“ und „Wahrscheinlichkeiten ableiten“ verwendet.
Leerer Konstruktor – Dieser Konstruktor akzeptiert keine Argumente und überlässt dem Benutzer die Parameter, die er später festlegen kann.
Ausführungskonstruktor – Dieser Konstruktor entfernt Argumente, die nur beobachtet werden, und verwendet für beobachtete Argumente, deren Dimensionen als Eingaben für den Code verwendet werden, diese Dimensionen anstelle der vollständigen Parameter. Im HMM-Beispiel wird der Parameter „eventsMeasured“ zu einer Ganzzahl, die die Länge der Sequenz beschreibt.
Diese Codebeispiele veranschaulichen, wie die kompilierten Modelle aufgerufen werden.
Interaktionen mit einem Modell über das Modellobjekt nehmen zwei Formen an:
Aufrufe von Modellobjektmethoden für globale Vorgänge wie das Festlegen von Standardaufbewahrungsrichtlinien, das Überprüfen, ob das Modell für die Inferenz bereit ist, das Starten von Inferenzschritten usw.
Aufrufe zum Modellieren von Parameterobjekten. Jede benannte öffentliche Variable im Modell wird durch ein entsprechendes Feld im Modellobjekt dargestellt. Variablen sind öffentlich, wenn sie im äußersten Bereich des Modells deklariert und nicht private gekennzeichnet sind, oder wenn sie in einem inneren Bereich deklariert sind und nicht public gekennzeichnet sind. Wenn ein Feld in einem inneren iterativen Bereich als öffentlich deklariert wird, beispielsweise im Rumpf einer for-Schleife, wird der Wert aus jeder Iteration gespeichert.
Der Typ des Objekts hängt von der Variablen ab. Diese können in 3 Kategorien unterteilt werden:
Jedes dieser Felder verweist auf ein Objekt mit einer Reihe von Methoden, die es dem Benutzer ermöglichen, Werte und Eigenschaften des Parameters festzulegen und zu lesen. Zu den Eigenschaften, die festgelegt und gelesen werden können, gehören die Wahrscheinlichkeit des Parameters, die Aufbewahrungsrichtlinie des Parameters und ob der Parameter auf seinen aktuellen Wert festgelegt werden soll.
Einige der wichtigeren Methoden des Parameterobjekts bei der Modellinferenz sind:
getSamples, um abgetastete Werte zurückzugeben.
getMAP, um den maximalen A-posteriori-Wert zurückzugeben.
setValue, um das Festlegen eines Werts auf einen bestimmten Wert zu ermöglichen.
setFixed, das einen boolean benötigt, um den Wert als fest zu markieren und daher während der Inferenz nicht aktualisiert zu werden. Es ist wichtig, den Wert des Parameters festzulegen, bevor Sie ihn korrigieren.
getLogProbability, das die Log-Wahrscheinlichkeit der Variablen nach Ableitung von Wahrscheinlichkeiten abruft.
Es gibt noch mehr Methoden und wir empfehlen, JavaDoc zu konsultieren, um sich mit ihnen vertraut zu machen.
Es gibt drei grundlegende Arten von Operationen, die an einem Modell durchgeführt werden können:
setRentionPolicy in der Modellklasse festgelegt werden. Optional kann dann die Aufbewahrungsrichtlinie für einzelne Variablen durch Aufrufe der entsprechenden setRetentionPolicy -Methode in jedem Variablenobjekt festgelegt werden.Es gibt drei Stichprobenrichtlinien:
NONE zeichnet keine Werte auf. Dies ist besonders nützlich, wenn eine der Variablen groß ist und der Zeit- und Platzbedarf für deren Speicherung daher verschwenderisch wäre.
SAMPLE zeichnet den Wert aus jeder Iteration des Inferenzalgorithmus auf. Wenn also 1000 Iterationen durchgeführt werden, werden 1000 Werte von jeder Variablen abgetastet, die auf diese Aufbewahrungsrichtlinie festgelegt ist. Dies ist sowohl für die Berechnung der Varianz als auch des Durchschnittswerts nützlich. Dies hat jedoch eine Schwäche: Wenn sich die Positionen der Werte innerhalb des Modells während der Inferenz verschieben können, kann über die Werte kein Mittelwert gebildet werden. Beispielsweise können bei einem Themenmodell die Themen 2 und 3 während der Inferenz die Plätze tauschen, so dass die Mittelung aller Werte für Thema 2 eine Mischung aus Thema 2 und Thema 3 ergibt. Um dieses Maximum A Posteriori (MAP) zu überwinden, wird auch ein Wert angegeben Aufbewahrungsrichtlinie.
MAP oder Maximum A Posteriori (MAP) zeichnet die Werte der Variablen auf, wenn sich das Modell in seinem wahrscheinlichsten Zustand befindet. Dadurch wird das Problem mit transienten Wertpositionen gelöst, was bedeutet, dass Werte nicht gemittelt werden können, allerdings auf Kosten der Möglichkeit, Grenzen zu berechnen. Diese Option bietet auch Platzvorteile, wenn einige der Variablen groß sind.
Konfiguration: Zusätzliche Methodenaufrufe für das Modellobjekt ermöglichen es dem Benutzer, Eigenschaften wie Burnin und Thinning festzulegen, wenn er diesen Inferenzschritt durchführt. Burnin ignoriert die Werte aus den ersten n Iterationen, sodass sich das Modell von einem Startpunkt mit geringer Wahrscheinlichkeit entfernen kann, bevor mit der Stichprobe begonnen wird. Durch die Ausdünnung wird die durch das MCMC-Verfahren induzierte Autokorrelation reduziert, indem nur die Werte aus jeder n -ten Iteration berücksichtigt werden.
Wahrscheinlichkeiten ableiten Nachdem Sie die Werte einiger oder aller Parameter im Modell festgelegt haben, berechnen Sie die Wahrscheinlichkeit der Generierung dieser Werte. Dies kann für jede Variable im Modell und für das Modell als Ganzes berechnet werden.
Modell ausführen Führen Sie das Modell aus, als wäre es normaler Code, der neue Werte für alle Parameter generiert, die nicht vom Benutzer festgelegt wurden. Ein Beispiel für den Einsatz dieses Verhaltens wäre ein lineares Regressionsmodell. In diesem Fall würden die Modellkoeffizienten zunächst mithilfe von Trainingsdaten abgeleitet. Sobald sie abgeleitet wurden, wären sie behoben und ein neuer Eingabedatensatz. Das Modell würde dann ausgeführt, um die entsprechenden Vorhersagen für diese neuen Eingabedaten zu generieren. Diese Ausführungsform könnte auch verwendet werden, um repräsentative synthetische Daten aus einem trainierten Modell zu generieren.
Konstruieren und trainieren Sie ein Modell
//Load inputs
int nStates = 25 ;
int [] actions = loadActions (....);
int nActions = maxActions (....);
//Construct the model
HMM model = new HMM ( actions , nActions , nStates );
//Set the retention policies
model . setDefaultRetentionPolicy ( RetentionPolicy . MAP );
model . st . setRetentionPolicy ( RetentionPolicy . NONE );
//Pick a random number generator. The ones introduced in Java 17 are faster and better quality.
model . setRNGType ( RandomType . L64X1024MixRandom );
//Instruct the model to use the ForkJoin framework for parallel execution.
model . setExecutionTarget ( ExecutionTarget . forkJoin );
//Run 2000 inference steps to infer model values
model . inferValues ( 2000 );
//Gather the results.
double [] initialState = model . initialState . getMAP ();
double [][] bias = model . bias . getMAP ();
double [][] transitions = model . m . getMAP ();Konstruieren Sie ein Modell und leiten Sie Wahrscheinlichkeiten ab
//Load inputs
int nStates = 25 ;
int [] actions = loadActions (....);
int nActions = maxActions (....);
//Load model parameters
double [][] bias = model . bias . getMAP ();
double [][] transitions = model . m . getMAP ();
//Construct the model
HMM model = new HMM ( actions , nActions , nStates );
//Set and fix trained values
model . bias . setValue ( bias );
Model . m . setValue ( transitions );
//Run 2000 inference steps to infer probabilities
model . inferProbabilities ( 2000 );
//Recover the probabilities of the model parameter actions.
double actionsProbability = model . actions . getProbability ();
//Recover the probability of the model as a whole
double modelProbability = model . getProbability ()Wenn Sie Hilfe zu Sandwood benötigen, starten Sie bitte eine Diskussion auf der Diskussionsseite oder nehmen Sie daran teil.
Dieses Projekt freut sich über Beiträge aus der Community. Bevor Sie eine Pull-Anfrage einreichen, lesen Sie bitte unseren Beitragsleitfaden.
Bitte konsultieren Sie den Sicherheitsleitfaden für unseren Prozess zur verantwortungsvollen Offenlegung von Sicherheitslücken.
Copyright (c) 2019–2024 Oracle und/oder seine verbundenen Unternehmen.
Veröffentlicht unter der Universal Permissive License v1.0, wie unter https://oss.oracle.com/licenses/upl/ gezeigt.