La implementación de DiffAbXL se compara en el artículo: Exploración de puntuaciones de probabilidad logarítmica para clasificar diseños de secuencias de anticuerpos.
| Rango | Modelos | Abscis HER2 | Naturaleza | Objetivo AZ-2 | Cra. ? | ||
|---|---|---|---|---|---|---|---|
| Tiro cero | Control de ESP | Hola | HER2 | ||||
| 1 | DiffAbXL-A-DN | 0,43 | 0,22 | 0,62 | 0,37 | 0,41 | 0,41 |
| 2 | DiffAbXL-A-SG | 0,46 | 0,22 | 0,64 | -0,38 | 0,43 | 0,274 |
| 3 | DiffAbXL-H3-DN | 0,49 | 0 | 0,52 | -0,08 | 0,37 | 0,26 |
| 4 | IgBlend (solo estructura) | 0,40 | 0,21 | 0,54 | -0,30 | 0,31 | 0,232 |
| 5 | Antipliegue | 0,43 | 0,22 | 0,4 | -0,47 | 0,38 | 0,192 |
| 6 | DiffAbXL-H3-SG | 0,48 | 0 | 0,4 | -0,41 | 0,29 | 0,152 |
| 7 | MEDE | 0,29 | 0 | 0 | 0,18 | 0,27 | 0,148 |
| 8 | DifAb | 0,34 | 0,21 | 0 | -0,14 | 0,22 | 0.126 |
| 9 | AbLang2 | 0.3 | 0 | 0 | -0,07 | 0,36 | 0.118 |
| 10 | IgBlend (solo secuencia) | 0,27 | 0 | 0 | -0,1 | 0,36 | 0.106 |
| 11 | AbLang | 0.3 | 0 | 0 | -0,13 | 0,35 | 0.104 |
| 12 | dyMEAN | 0,37 | 0,15 | 0 | 0 | 0 | 0.104 |
| 13 | Abx | 0,28 | 0,19 | 0 | 0 | 0 | 0.094 |
| 14 | AntiBERTY | 0,26 | 0 | 0 | -0,17 | 0,35 | 0,088 |
| 15 | SIGNIFICAR | 0,36 | 0 | 0 | 0,02 | 0 | 0,076 |
| 16 | MEDE-SI | 0 | -0,27 | 0 | -0,53 | 0,42 | -0,076 |
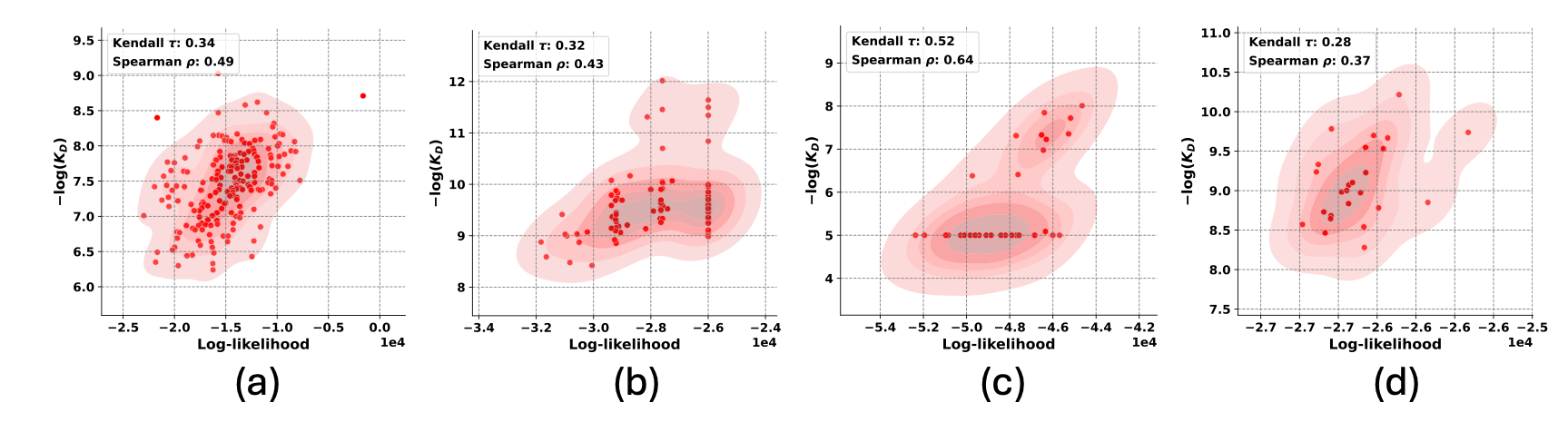
Figura-1: Resultados para DiffAbXL: a) DiffAbXL-H3-DN para datos Absci zero-shot HER2 b) DiffAbXL-A-SG para AZ Target-2, c) DiffAbXL-A-SG para Nature HEL, d) DiffAbXL- A-DN para la naturaleza HER2.
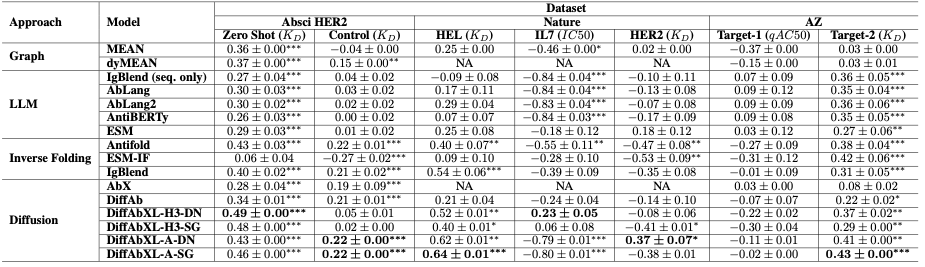
Tabla-1: Resumen de los resultados de la correlación de Spearman. Abreviaturas: DN: modo De Novo, SG: modo de guía de estructura, NA: epítopo o estructura compleja requerida, pero no disponible. *, **, *** indican valores de p inferiores a 0,05, 0,01 y 1e-4 respectivamente.
Para que nos resulte más fácil comparar su modelo, le recomendamos que implemente una interfaz como método de Python en una clase que podamos integrar fácilmente con nuestro proceso de evaluación. El método debe aceptar las siguientes entradas:
El método debería devolver un diccionario que contenga:
Aquí hay una plantilla básica en Python para implementar esta interfaz:
def benchmark ( self , sequences , structure = None , mask = None , ** kwargs ):
"""
Benchmark the model on provided antibody sequences and structures.
Parameters:
sequences (list of str): List of antibody sequences.
structure (optional): Path to a PDB file. Currently, only one PDB file is provided per target dataset.
The PDB file may contain either just the antibody or an antibody-antigen complex,
depending on the dataset.
mask (optional): Binary list or array indicating the regions of interest in the sequences for metric calculations.
kwargs (optional): Additional parameters required by the model.
Returns:
dict: A dictionary containing log-likelihood scores and other relevant metrics.
"""
passAsegúrese de que su modelo genere las puntuaciones de probabilidad logarítmica en un formato que podamos usar directamente para comparar diseños de secuencias de anticuerpos. Esto nos ayudará a comparar el rendimiento de su modelo en nuestros conjuntos de datos de manera eficiente.
Hay un archivo de configuración: sabdab.yaml, que se puede utilizar para cambiar cualquiera de los parámetros. Puedes entrenar el modelo usando:
python train.py # For training.
- tren.py
-src
|-modelo.py
- configuración
|-sabdab.yaml
- utiles
|-load_data.py
|-argumentos.py
|-model_utils.py
|-loss_functions.py
...
- datos
|-her2
...
Weight & Biases se puede utilizar para realizar un seguimiento de los experimentos. Está desactivado de forma predeterminada, pero se puede activar cambiando la opción en el archivo de configuración en ./config/sabdab.yaml
@article {Ucar2024.10.07.617023,
author = {Ucar, Talip and Malherbe, Cedric and Gonzalez Hernandez, Ferran},
title = {Exploring Log-Likelihood Scores for Ranking Antibody Sequence Designs},
elocation-id = {2024.10.07.617023},
year = {2024},
doi = {10.1101/2024.10.07.617023},
publisher = {Cold Spring Harbor Laboratory},
URL = {https://www.biorxiv.org/content/early/2024/10/24/2024.10.07.617023},
eprint = {https://www.biorxiv.org/content/early/2024/10/24/2024.10.07.617023.full.pdf},
journal = {bioRxiv}
}
Si utiliza DiffAbXL en sus propios estudios y trabajo, cítelo utilizando lo siguiente:
@Misc{talip_ucar_2024_DiffAbXL,
author = {Talip Ucar},
title = {Exploring Log-Likelihood Scores for Ranking Antibody Sequence Designs},
URL = {https://github.com/AstraZeneca/DiffAbXL},
month = {October},
year = {since 2024}
}