AlphaFold3 (AF3), récemment développé par l'équipe DeepMind, a réalisé des progrès révolutionnaires dans le domaine de la prédiction de la structure des protéines. Il peut non seulement prédire la structure d'une seule protéine, mais également prédire la structure de complexes protéiques, d'acides nucléiques ou de petites molécules. . Les éditeurs de downcodes vous donneront une compréhension approfondie du fonctionnement interne d'AF3 et de la manière dont il accomplit cette tâche complexe grâce à une architecture intelligente. AF3 est comme un « boulanger » expérimenté qui « cuit » avec précision la structure tridimensionnelle de la protéine selon la « recette » fournie (séquence protéique). Le processus est comme dessiner une belle peinture, avec des couches superposées et finalement présentées. la structure complète.
AlphaFold3, appelé AF3, est le dernier chef-d'œuvre de l'équipe DeepMind dans le domaine de la prédiction de la structure des protéines. Il est capable de prédire la structure non seulement de séquences protéiques individuelles, mais également de complexes protéiques, d'acides nucléiques ou de petites molécules. C'est comme si vous donniez à AF3 une « recette » pour une protéine, il peut « cuire » la structure tridimensionnelle de la protéine.
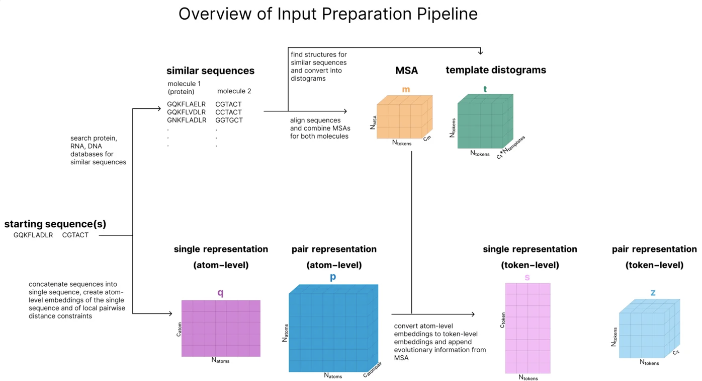
L’architecture d’AF3 est complexe et subtile, mais n’ayez crainte, une image peut vous aider à faire le tri. L'ensemble du modèle peut être divisé en trois parties :
Préparation des entrées : convertissez des séquences protéiques en tenseurs numériques et récupérez des molécules avec des structures similaires.
Apprentissage des représentations : utiliser plusieurs mécanismes d'attention pour mettre à jour ces représentations.
Prédiction de structure : prédisez les structures protéiques à l’aide de modèles de diffusion conditionnelle.
Chaque étape est comme dessiner une peinture délicate. AF3 affiche enfin la structure tridimensionnelle de la protéine couche par couche.
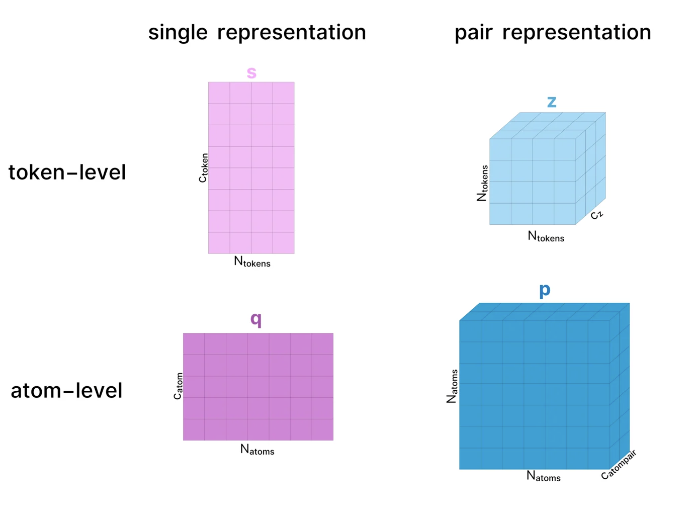
Dans le monde d'AF3, chaque molécule possède son propre « langage ». Qu'il s'agisse de protéines, d'ADN, d'ARN ou de petites molécules, AF3 peut les convertir en une série de tenseurs numériques. C'est comme donner à chaque molécule un « ID » unique qui permet à AF3 de les reconnaître et de les traiter.
La partie apprentissage de la représentation d’AF3 est comme une danse soigneusement chorégraphiée. Grâce au mécanisme d'attention, AF3 peut laisser la « vue » du modèle se promener entre différentes parties de la molécule et capturer la relation entre elles. Cela inclut non seulement les interactions au sein des molécules, mais également les interactions entre molécules.
Dans la partie prédiction de structure d’AF3, le modèle de diffusion conditionnelle joue un rôle clé. Cela commence par une série de bruits aléatoires et les « débruit » progressivement, rétablissant finalement la véritable structure de la protéine. Ce processus revient à révéler progressivement la vérité cachée d’un brouillard.
La formation d'AF3 implique une variété de fonctions de perte et de têtes de confiance, qui fonctionnent ensemble pour permettre à AF3 de prédire les structures avec plus de précision et d'évaluer la fiabilité de ses prédictions. C'est comme mettre un miroir sur AF3, lui permettant de se refléter et de s'améliorer.
Référence : https://elanapearl.github.io/blog/2024/the-illustrated-alphafold/
Dans l’ensemble, AlphaFold3 a apporté des changements révolutionnaires dans le domaine de la prédiction de la structure des protéines grâce à son architecture exquise et ses puissantes capacités d’apprentissage. Il a de larges perspectives d’application et devrait jouer un rôle majeur dans la biomédecine, la science des matériaux et d’autres domaines. J'espère que l'explication de l'éditeur de Downcodes pourra vous aider à mieux comprendre cette technologie étonnante.