Repo ini berisi:
sepal membutuhkan python3 , sebaiknya versi yang lebih baru atau sama dengan 3.5. Untuk mengunduh dan menginstal, buka terminal dan ubah ke direktori tempat Anda ingin sepal diunduh dan lakukan:
git clone https://github.com/almaan/sepal.git
cd sepal
chmod +x setup.py
./setup.py install
Bergantung pada hak istimewa pengguna, Anda mungkin harus menambahkan --user sebagai argumen ke setup.py . Menjalankan pengaturan akan memberi Anda instalasi minimal yang diperlukan untuk menghitung waktu difusi. Namun, jika Anda ingin dapat menggunakan modul analisis, Anda juga perlu menginstal paket yang direkomendasikan. Untuk melakukan ini, cukup jalankan (dalam direktori yang sama):
pip install -e " .[full] " sekali lagi, --user mungkin perlu disertakan. Selain itu, Anda mungkin harus menggunakan pip3 jika ini adalah cara Anda mengatur antarmuka python-pip Anda. Jika Anda menggunakan conda atau lingkungan virtual, ikuti rekomendasi mereka untuk instalasi paket.
Ini harus menginstal antarmuka baris perintah (CLI) dan paket standar. Untuk menguji dan melihat apakah instalasi berhasil Anda dapat mencoba menjalankan perintah:
sepal -h
Yang seharusnya mencetak pesan bantuan yang terkait dengan sepal. Jika sejauh ini semuanya berhasil, Anda dapat melanjutkan ke bagian contoh untuk melihat sepal beraksi!
Penggunaan sepal yang disarankan adalah melalui antarmuka baris perintah. Baik simulasi untuk menghitung waktu difusi serta analisis atau pemeriksaan hasil selanjutnya dapat dengan mudah dilakukan dengan mengetikkan sepal diikuti dengan run atau analyze . Modul analyze memiliki opsi yang berbeda, untuk memvisualisasikan hasil ( inspect ), mengurutkan profil ke dalam kelompok pola ( family ) atau mengarahkan keluarga yang teridentifikasi ke analisis pengayaan fungsional ( fea ). Untuk daftar lengkap perintah yang tersedia, lakukan sepal module -h , di mana module adalah salah satu run dan analyze . Di bawah ini, kami mengilustrasikan bagaimana sepal dapat digunakan untuk menemukan profil transkripsi dengan pola spasial.
Kami akan membuat folder untuk menyimpan hasil kami, yang juga akan berfungsi sebagai direktori kerja kami. Dari direktori utama repo, lakukan:
cd res
mkdir example
cd exampleSampel MOB akan digunakan sebagai contoh analisis kami. Kami mulai dengan menghitung waktu difusi untuk setiap profil transkripsi:
sepal run -c ../../data/real/mob.tsv.gz -mo 10 -mc 5 -o . -ar 1Di bawah ini adalah contoh (dengan tampilan tambahan dari perintah bantuan) bagaimana tampilannya

Setelah menghitung waktu difusi, kami ingin memeriksa hasilnya, seperti dalam penelitian ini, kami akan melihat 20 profil teratas. Kita dapat dengan mudah menghasilkan gambar dari hasil kita dengan menjalankan perintah:
sepal analyze -c ../../data/real/mob.tsv.gz
-r 20200409173043610345-top-diffusion-times.tsv
-ar 1k -o . inspect -ng 20 -nc 5Yang akan terlihat seperti ini:

Outputnya akan menjadi gambar berikut:
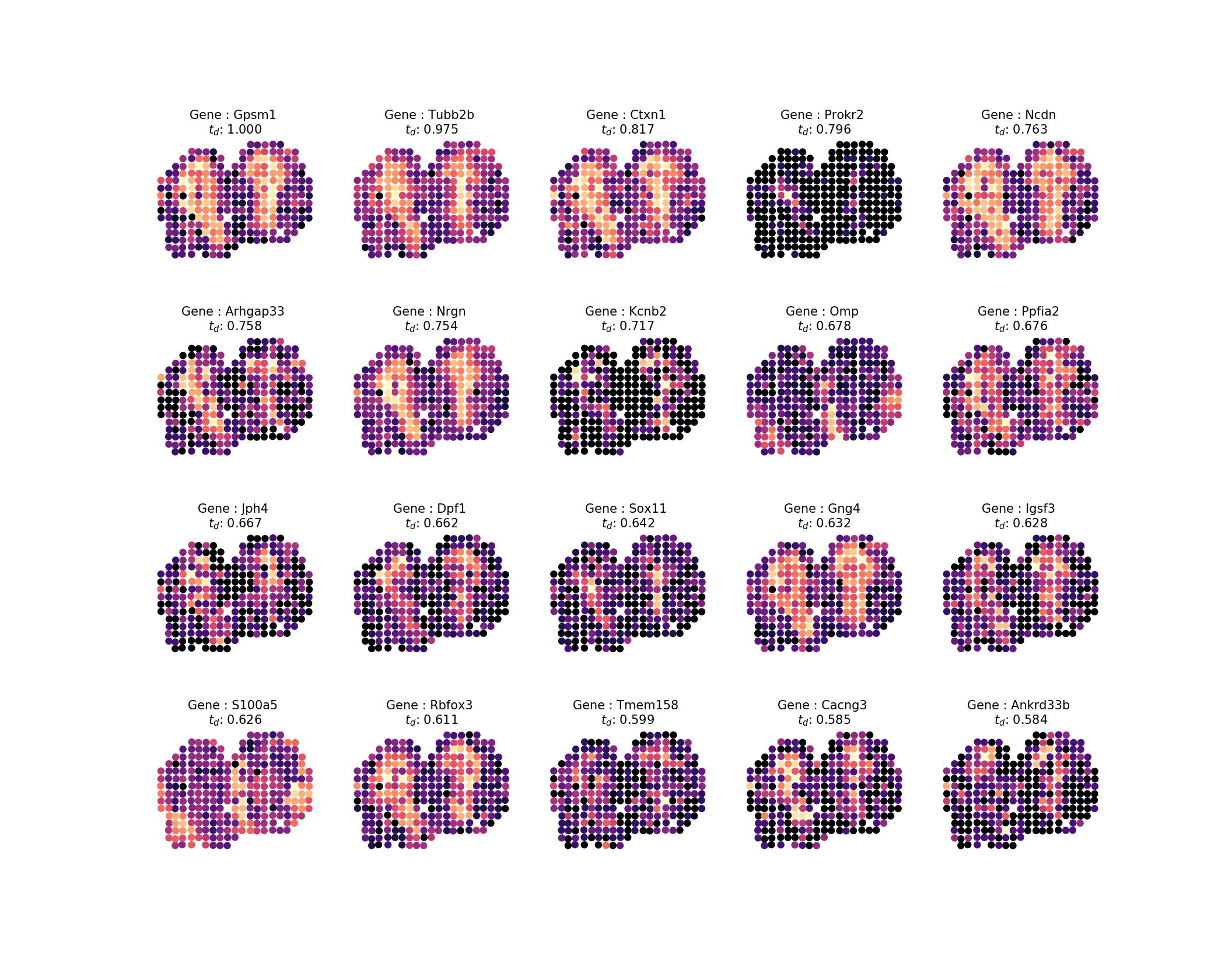
Kemudian, untuk mengurutkan 100 gen peringkat teratas ke dalam serangkaian keluarga pola, yang mana 85% varian dalam pola kita harus dijelaskan oleh pola eigen, lakukan:
sepal analyze -c ../../data/real/mob.tsv.gz
-r 20200409173043610345-top-diffusion-times.tsv
-ar 1k -o . family -ng 100 -nbg 100 -eps 0.85 --plot -nc 3Dari sini diperoleh tiga motif yang mewakili setiap keluarga sebagai berikut:
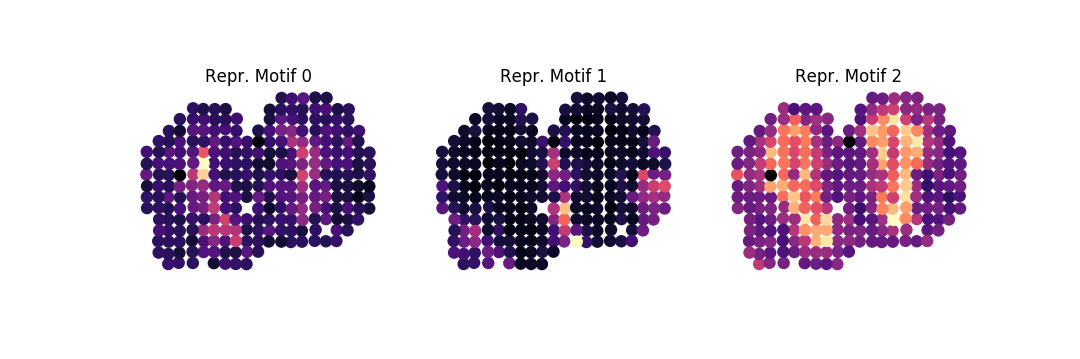
Kita dapat melakukan analisis pengayaan pada keluarga kita, dengan menjalankan:
sepal analyze -c ../../data/real/mob.tsv.gz
-r 20200409173043610345-top-diffusion-times.tsv
-ar 1k -o . fea -fl mob.tsv-family-index.tsv -or " mmusculus "di mana kita misalnya melihat bahwa Keluarga 2 diperkaya untuk beberapa proses yang berkaitan dengan fungsi, pembangkitan, dan regulasi saraf:
| keluarga | warga asli | nama | nilai_p | sumber | ukuran_persimpangan | |
|---|---|---|---|---|---|---|
| 2 | 2 | PERGI:0007399 | perkembangan sistem saraf | 0,00035977 | PERGI:BP | 26 |
| 3 | 2 | PERGI:0050773 | regulasi perkembangan dendrit | 0,000835883 | PERGI:BP | 8 |
| 4 | 2 | PERGI:0048167 | regulasi plastisitas sinaptik | 0,00196494 | PERGI:BP | 8 |
| 5 | 2 | PERGI:0016358 | pengembangan dendrit | 0,00217167 | PERGI:BP | 9 |
| 6 | 2 | PERGI:0048813 | morfogenesis dendrit | 0,00741589 | PERGI:BP | 7 |
| 7 | 2 | PERGI:0048814 | regulasi morfogenesis dendrit | 0,00800399 | PERGI:BP | 6 |
| 8 | 2 | PERGI:0048666 | perkembangan neuron | 0,0114088 | PERGI:BP | 16 |
| 9 | 2 | PERGI:0099004 | jalur pensinyalan kinase dependen calmodulin | 0,0159572 | PERGI:BP | 3 |
| 10 | 2 | PERGI:0050804 | modulasi transmisi sinaptik kimia | 0,0341913 | PERGI:BP | 10 |
| 11 | 2 | PERGI:0099177 | regulasi sinyal trans-sinaptik | 0,0347783 | PERGI:BP | 10 |
Tentu saja analisis ini tidak menyeluruh. Melainkan contoh singkat untuk menunjukkan bagaimana seseorang mengoperasikan CLI untuk sepal .
Meskipun sepal telah dirancang sebagai alat mandiri, kami juga membuatnya berfungsi sebagai paket python standar yang fungsinya dapat diimpor dan digunakan dalam alur kerja terintegrasi. Untuk menunjukkan bagaimana hal ini dapat dilakukan, kami memberikan contoh, mereproduksi analisis melanoma. Contoh lainnya dapat ditambahkan nanti.
Input ke sepal harus dalam format n_locations x n_genes , namun jika data Anda terstruktur dengan cara yang berlawanan ( n_genes x n_locations ) cukup berikan tanda --transpose saat menjalankan simulasi atau analisis dan ini akan ditangani dari.
Saat ini kami mendukung format .csv , .tsv dan .h5ad . Untuk yang terakhir, file Anda harus disusun menurut format INI. Kami berharap dalam waktu dekat akan ada rilis dari tim scanpy yang menyajikan format standar untuk data spasial, namun hingga saat itu kami akan menggunakan standar tersebut di atas.
Semua data nyata yang kami gunakan bersifat publik, dan dapat diakses di tautan berikut:
Data sintetis dihasilkan oleh:
synthetic/img2cnt.pysynthetic/turing.pysynthetic/ablation.py Semua hasil penelitian yang disajikan dapat ditemukan di folder res , baik untuk data nyata maupun sintetik. Untuk setiap sampel, kami telah menyusun hasilnya sesuai dengan:
res/sample-name/X-diffusion-times.tsv : waktu difusi untuk semua gen yang diberi peringkatanalysis/ : berisi keluaran analisis sekunder