Fantastisches KI-basiertes Proteindesign
Dies ist eine Sammlung von Forschungsarbeiten zum KI-basierten Proteindesign . Und das Repository wird kontinuierlich aktualisiert, um die Grenzen des KI-basierten Proteindesigns zu verfolgen.
Willkommen zum Folgen und Starren!
Inhaltsverzeichnis
Überblick über Proteindesign
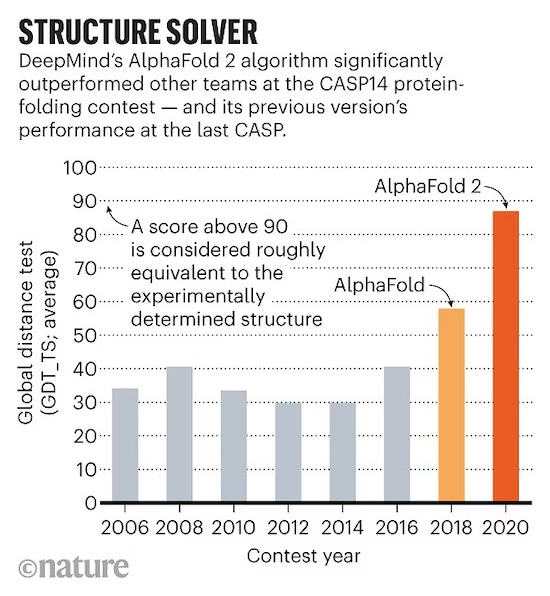
KI-Tools haben das Problem der Proteinstrukturvorhersage gelöst. Dieses Problem leitet die räumliche Struktur aus der Aminosäuresequenz ab und erreicht eine Vorhersagegenauigkeit auf atomarer Ebene, wie z. B. AlphaFold 2. Es kombiniert frühere Proteinstruktur-Vorhersagemodelle, um automatisch Proteindesignmethoden zu erlernen und so den pharmazeutischen Bedürfnissen des Menschen wirklich gerecht zu werden.
Die spezifischen Praktiken des Proteindesigns variieren stark, und auch die Problemdefinitionen, die auf verschiedene Designprozesse anwendbar sind, sind sehr unterschiedlich. Hier einige Beispiele:
- Ein Problem der Vorhersage von Aminosäuresequenzen anhand der räumlichen Struktur (die Umkehrung von Alphafold), bei dem davon ausgegangen wird, dass die räumliche Struktur des gewünschten Proteins durch Molekulardynamiksimulationen usw. abgeleitet werden kann.
- Ein Problem der Vervollständigung der Proteinstruktur für eine bestimmte Teilstruktur, wie beispielsweise das aktuelle Science [1] der berühmten David Baker-Gruppe. Dabei wird davon ausgegangen, dass nur teilweise strukturelle Übereinstimmungen gefunden werden können.
- Kombination der angepassten Energiefunktion mit MD-Simulation für das Proteindesign, wie beispielsweise die aktuelle Nature [2] des Teams von Liu Haiyan in China.
Darüber hinaus können viele Methoden für das Proteindesign verwendet werden, und auch die entsprechenden KI-Problemdefinitionen sind sehr unterschiedlich. Dieser Artikel listet einige hochrangige Artikel zum KI-basierten Proteindesign auf, die in Zukunft kontinuierlich aktualisiert werden.
Papiere
format:
- [title](paper link) [links]
- author1, author2, and author3...
- publisher
- keyword
Natur
Genaue Strukturvorhersage biomolekularer Wechselwirkungen mit AlphaFold 3
- Josh Abramson, Jonas Adler, Jack Dunger, Richard Evans, Tim Green, Alexander Pritzel, Olaf Ronneberger, Lindsay Willmore, Andrew J. Ballard, Joshua Bambrick, Sebastian W. Bodenstein, David A. Evans, Chia-Chun Hung, Michael O' Neill, David Reiman, Kathryn Tunyasuvunakool, Zachary Wu, Akvilė Žemgulytė, Eirini Arvaniti, Charles Beattie, Ottavia Bertolli, Alex Bridgland, Alexey Cherepanov, Miles Congreve, Alexander I. Cowen-Rivers, Andrew Cowie, Michael Figurnov, Fabian B. Fuchs, Hannah Gladman, Rishub Jain, Yousuf A. Khan, Caroline MR Low, Kuba Perlin, Anna Potapenko , Pascal Savy, Sukhdeep Singh, Adrian Stecula, Ashok Thillaisundaram, Catherine Tong, Sergei Yakneen, Ellen D. Zhong, Michal Zielinski, Augustin Žídek, Victor Bapst, Pushmeet Kohli, Max Jaderberg, Demis Hassabis und John M. Jumper
- Schlüsselwort: Diffusionsbasierte Architektur, Proteinstrukturmodellierung, biomolekulare Raummodellierung
Eine rückgratzentrierte Energiefunktion neuronaler Netze für das Proteindesign
- B. Huang, Y. Xu, X. Hu, Y. Liu, S. Liao, J. Zhang, C. Huang
- Stichwort: Energiefunktion, MD-Simulation, rückgratzentriert
De-novo-Proteindesign durch tiefe Netzwerkhalluzination
- Ivan Anishchenko, Samuel J. Pellock, Tamuka M. Chidyausiku, Theresa A. Ramelot, Sergey Ovchinnikov, Jingzhou Hao, Khushboo Bafna, Christoffer Norn, Alex Kang, Asim K. Bera, Frank DiMaio, Lauren Carter, Cameron M. Chow, Gaetano T. Montelione & David Baker
- Schlüsselwörter: Halluzination, Inpainting, Proteindesign
Design proteinbindender Proteine allein aus der Zielstruktur
- Longxing Cao, Brian Coventry, Inna Goreshnik, Buwei Huang, William Sheffler, Joon Sung Park, Kevin M. Jude, Iva Marković, Rameshwar U. Kadam, Koen HG Verschueren, Kenneth Verstraete, Scott Thomas Russell Walsh, Nathaniel Bennett, Ashish Phal, Aerin Yang, Lisa Kozodoy, Michelle DeWitt, Lora Picton, Lauren Miller, Eva-Maria Strauch, Nicholas D. DeBouver, Allison Pires, Asim K. Bera, Samer Halabiya, Bradley Hammerson, Wei Yang, Steffen Bernard, Lance Stewart, Ian A. Wilson, Hannele Ruohola-Baker, Joseph Schlessinger, Sangwon Lee, Savvas N. Savvides, K. Christopher Garcia & David Baker
- Schlüsselwörter: verbindliches Zitat
Naturbiomedizinische Technik
- Beschleunigte antimikrobielle Entdeckung durch tiefgreifende generative Modelle und Molekulardynamiksimulationen
- Payel Das, Tom Sercu, Kahini Wadhawan, Inkit Padhi, Sebastian Gehrmann, Flaviu Cipcigan, Vijil Chenthamarakshan, Hendrik Strobelt, Cicero dos Santos, Pin-Yu Chen, Yi Yan Yang, Jeremy PK Tan, James Hedrick, Jason Crain und Aleksandra Mojsilovic
- Schlüsselwörter: antimikrobielle Mittel, generativer Autoencoder, Molekulardynamik
Naturkommunikation
Entdeckung von De-novo-Peptidsubstraten für Enzyme mithilfe maschinellen Lernens
- Lorillee Tallorin, JiaLei Wang, Woojoo E. Kim, Swagat Sahu, Nicolas M. Kosa, Pu Yang, Matthew Thompson, Michael K. Gilson, Peter I. Frazier, Michael D. Burkart und Nathan C. Gianneschi
- Schlüsselwörter: Enzymdesign, maschinelles Lernen
ECNet ist ein evolutionäres, kontextintegriertes Deep-Learning-Framework für Protein-Engineering
- Yunan Luo, Guangde Jiang, Tianhao Yu, Yang Liu, Lam Vo, Hantian Ding, Yufeng Su, Wesley Wei Qian, Huimin Zhao und Jian Peng
- Schlüsselwörter: funktionelle Fitness, evolutionär
Proteindesign und Variantenvorhersage mithilfe autoregressiver generativer Modelle
- Jung-Eun Shin, Adam J. Riesselman, Aaron W. Kollasch, Conor McMahon, Elana Simon, Chris Sander, Aashish Manglik, Andrew C. Kruse und Debora S. Marks
- Schlüsselwörter: autoregressive generative Modelle, Proteindesign
Proteinsequenzdesign mit erlerntem Potenzial
- Namrata Anand, Raphael Eguchi, Irimpan I. Mathews, Carla P. Perez, Alexander Derry, Russ B. Altman und Po-Ssu Huang
- Schlüsselwörter: Proteindesign, Energiefunktion, tiefes neuronales Netzwerk
Proteindesign und Variantenvorhersage mithilfe autoregressiver generativer Modelle
- Jung-Eun Shin, Adam J. Riesselman, Aaron W. Kollasch, Conor McMahon, Elana Simon, Chris Sander, Aashish Manglik, Andrew C. Kruse und Debora S. Marks
- Schlüsselwörter: autoregressive generative Modelle, Proteindesign
Naturmaschinenintelligenz
Wissenschaft
Robustes, auf Deep Learning basierendes Proteinsequenzdesign mit ProteinMPNN
- J. Dauparas, I. Anishchenko, N. Bennett, H. Bai, RJ Ragotte, LF Milles, BIM Wicky, A. Courbet, RJ de Haas, N. Bethel, PJY Leung, TF Huddy, S. Pellock, D. Tischer , F. Chan, B. Koepnick, H. Nguyen, A. Kang, B. Sankaran, AK Bera, NP King, D. Baker
- Schlüsselwörter: Sprachmodell, Strukturvorhersage
Aufbau von Proteinfunktionsstellen mithilfe von Deep Learning
- Jue Wang, Sidney Lisanza, David Jürgens, Doug Tischer, Joseph L. Watson, Karla M. Castro, Robert Ragotte, Amijai Saragovi, Lukas F. Milles, Minkyung Baek, Ivan Anishchenko, Wei Yang, Derrick R. Hicks, Marc Expòsit, Thomas Schlichthaerle, Jung-Ho Chun, Justas Dauparas, Nathaniel Bennett, Basile IM Wicky, Andrew Muenks, Frank DiMaio, Bruno Correia, Sergey Ovchinnikov, David Baker
- Schlüsselwörter: Funktionsstandort, Deep Learning, Halluzination, Inpainting
ICML, ICLR oder NeurIPS
Deep Reinforcement Learning zur Modellierung von Proteinkomplexen
- Ziqi Gao, Tao Feng, Jiaxuan You, Chenyi Zi, Yan Zhou, Chen Zhang, Jia Li
- Schlüsselwörter: Vorhersage komplexer Proteinstrukturen, Vorhersage des Andockpfads, Richtliniennetzwerk, Verstärkungslernen
BERTologie trifft Biologie: Aufmerksamkeit in Proteinsprachmodellen interpretieren
- Jesse Vig, Ali Madani, Lav R. Varshney, Caiming Xiong, Richard Socher, Nazneen Fatema Rajani
- Schlüsselwörter: Sprachmodell, Transformator, strukturelle und funktionale Eigenschaft
Bedingtes Antikörperdesign als äquivariante 3D-Graphtranslation
- Xiangzhe Kong, Wenbing Huang, Yang Liu
- Schlüsselwörter: Antikörperdesign, Diagrammübersetzung
Konditionierung durch adaptives Sampling für robustes Design
- David Brookes, Hahnbeom Park, Jennifer Listgarten
- Schlüsselwörter: Adaptive Probenahme, Proteindesign
Tiefe generative Modelle erzeugen neue und vielfältige Proteinstrukturen
- Zeming Lin, Tom Sercu, Yann LeCun
- Schlüsselwörter: Diversität, generatives Modell, Proteindesign
Tiefgreifende Schärfung topologischer Merkmale für das De-novo-Proteindesign
- Zander Harteveld, Joshua Southern, Michaël Defferrard, Andreas Loukas, Pierre Vandergheynst, Micheal Bronstein, Bruno Correia
- Schlüsselwörter: Variations-Autoencoder, topologische Merkmale, Schärfen
Fold2Seq: Ein auf Joint Sequence(1D)-Fold(3D)-Einbettung basierendes generatives Modell für das Proteindesign
- Yue Cao, Payel Das, Vijil Chenthamarakshan, Pin-Yu Chen, Igor Melnyk, Yang Shen
- Schlüsselwörter: generatives Modell, Proteindesign
Generative Modellierung für Proteinstrukturen
- Namrata Anand, Possu Huang
- Schlüsselwörter: generatives Modell, Proteindesign
Generative Modelle für graphbasiertes Proteindesign
- John Ingraham, Vikas Garg, Regina Barzilay, Tommi Jaakkola
- Schlüsselwörter: generatives Modell, Proteindesign
Modellbasiertes Verstärkungslernen für das Design biologischer Sequenzen
- XChristof Angermueller, David Dohan, David Belanger, Ramya Deshpande, Kevin Murphy, Lucy Colwell
- Schlüsselwörter: Reinforcement Learning, Sequenzdesign
Molekülerzeugung für die Bindung von Zielproteinen mit Strukturmotiven
- Zaixi Zhang, Yaosen Min, Shuxin Zheng, Qi Liu
- Schlüsselwörter: Zielprotein, Strukturmotive, Fragment-für-Fragment-Generierung
Lernen der Proteinrepräsentation durch Vortraining mit geometrischer Struktur
- Zuobai Zhang, Minghao Xu, Arian Jamasb, Vijil Chenthamarakshan, Aurelie Lozano, Payel Das, Jian Tang
- Schlüsselwörter: Arzneimittelentwicklung, Arzneimitteldesign, generative Modelle neuer molekularer Strukturen
Arxiv oder bioRxiv
Proteinerzeugung mit evolutionärer Diffusion: Sequenz ist alles, was Sie brauchen
- Sarah Alamdari, Nitya Thakkar, Rianne van den Berg, Alex Xijie Lu, Nicolo Fusi, Ava Pardis Amini, Kevin K Yang
- Schlüsselwörter: Diffusionsmodell, tiefes generatives Modell, Proteingenerierung, Framework, Sequenzdesign
Eine High-Level-Programmiersprache für generatives Proteindesign
- Brian Hie, Salvatore Candido, Zeming Lin, Ori Kabeli, Roshan Rao, Nikita Smetanin, Tom Sercu, Alexander Rives
- Schlüsselwörter: ESMFold, Sprachmodell, energiebasiert
Breit anwendbares und genaues Proteindesign durch Integration von Strukturvorhersagenetzwerken und generativen Diffusionsmodellen
- Joseph L. Watson, David Jürgens, Nathaniel R. Bennett, Brian L. Trippe, Jason Yim, Helen E. Eisenach, Woody Ahern, Andrew J. Borst, Robert J. Ragotte, Lukas F. Milles, Basile IM Wicky, Nikita Hanikel , Samuel J. Pellock, Alexis Courbet, William Sheffler, Jue Wang, Preetham Venkatesh, Isaac Sappington, Susana Vázquez Torres, Anna Lauko, Valentin De Bortoli, Emile Mathieu, Regina Barzilay, Tommi S. Jaakkola, Frank DiMaio, Minkyung Baek, David Baker
- Schlüsselwörter: Verbreitung, allgemeines Deep-Learning-Framework, De-novo-Binder-Design
Funktionsgesteuertes Proteindesign durch tiefe Mannigfaltigkeitsprobenahme
- Vladimir Gligorijević, Daniel Berenberg, Stephen Ra, Simon Kelow, Kyunghyun Cho
- Schlüsselwörter: Sequenzentrauschender Autoencoder, tiefe Mannigfaltigkeitsabtastung
Sprachmodelle verallgemeinern über natürliche Proteine hinaus
- Robert Verkuil, Ori Kabeli, Yilun Du, Basile IM Wicky, Lukas F. Milles, Justas Dauparas, David Baker, Sergey Ovchinnikov, Tom Sercu, Alexander Rives
- Schlüsselwörter: ESMFold, Sprachmodell, festes Backbone-Design
Sprachmodelle von Proteinsequenzen auf der Skala der Evolution ermöglichen eine genaue Strukturvorhersage
- Zeming Lin, Halil Akin, Roshan Rao, Brian Hie, Zhongkai Zhu, Wenting Lu, Allan dos Santos Costa, Maryam Fazel-Zarandi, Tom Sercu, Sal Candido, Alexander Rives
- Schlüsselwörter: Strukturvorhersage, Sprachmodell
TERMinator: Ein neuronales Framework für strukturbasiertes Proteindesign unter Verwendung tertiärer Wiederholungsmotive
- Alex J. Li, Vikram Sundar, Gevorg Grigoryan, Amy E. Keating
- Schlüsselwörter: Proteindesign, Tertiärmotive
Sprachmodelle verallgemeinern über natürliche Proteine hinaus
- Robert Verkuil, Ori Kabeli, Yilun Du, Basile IM Wicky, Lukas F. Milles, Justas Dauparas, David Baker, Sergey Ovchinnikov, Tom Sercu, Alexander Rives
- Schlüsselwörter: Sprachmodell, Proteindesign
Andere
Referenz
[1] Wang, Jue et al. „Gerüst für funktionelle Proteinstandorte mithilfe von Deep Learning.“ Wissenschaft 377.6604 (2022): 387-394.
[2] Huang, Bin et al. „Eine rückgratzentrierte Energiefunktion neuronaler Netze für das Proteindesign.“ Natur 602.7897 (2022): 523-528.
Mitwirken
Unser Ziel ist es, dieses Repo noch besser zu machen. Wenn Sie daran interessiert sind, einen Beitrag zu leisten, finden Sie HIER Anweisungen zum Beitragen.
Lizenz
Das fantastische KI-basierte Proteindesign wird unter der Apache 2.0-Lizenz veröffentlicht.

