Impresionante diseño de proteínas basado en IA
Esta es una colección de artículos de investigación sobre diseño de proteínas basado en IA . Y el repositorio se actualizará continuamente para rastrear la frontera del diseño de proteínas basado en IA.
¡Bienvenido a seguir y protagonizar!
Tabla de contenido
Descripción general del diseño de proteínas
Las herramientas de inteligencia artificial han resuelto el problema de predicción de la estructura de las proteínas. Este problema deriva la estructura espacial de la secuencia de aminoácidos y logra una precisión de predicción a nivel atómico, como AlphaFold 2. Combina modelos de predicción de la estructura de proteínas anteriores para aprender automáticamente métodos de diseño de proteínas, satisfaciendo así verdaderamente las necesidades farmacéuticas humanas.
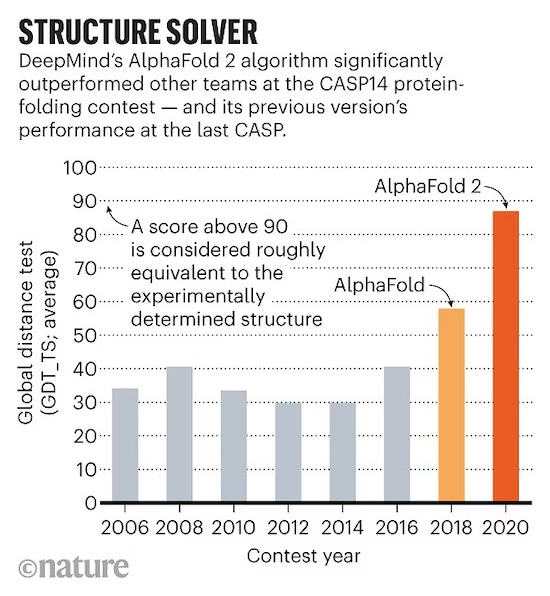
Las prácticas específicas del diseño de proteínas varían ampliamente y las definiciones de problemas aplicables a diferentes procesos de diseño también son muy diferentes. A continuación se muestran algunos ejemplos:
- Un problema de predicción de secuencias de aminoácidos a partir de la estructura espacial (lo inverso de Alphafold), que supone que la estructura espacial de la proteína deseada se puede derivar mediante simulaciones de dinámica molecular, etc.
- Un problema de finalización de la estructura de proteínas para una estructura parcial dada, como el reciente Science [1] del famoso grupo de David Baker. Esto supone que sólo se pueden encontrar coincidencias estructurales parciales.
- Combinación de la función de energía ajustada con simulación MD para el diseño de proteínas, como el reciente Nature [2] del equipo de Liu Haiyan en China.
Además, se pueden utilizar muchos métodos para el diseño de proteínas y las definiciones de problemas de IA correspondientes también son muy diferentes. Este artículo enumera algunos artículos de alto nivel sobre diseño de proteínas basado en IA, que se actualizarán continuamente en el futuro.
Papeles
format:
- [title](paper link) [links]
- author1, author2, and author3...
- publisher
- keyword
Naturaleza
Predicción estructural precisa de interacciones biomoleculares con AlphaFold 3
- Josh Abramson, Jonas Adler, Jack Dunger, Richard Evans, Tim Green, Alexander Pritzel, Olaf Ronneberger, Lindsay Willmore, Andrew J. Ballard, Joshua Bambirick, Sebastian W. Bodenstein, David A. Evans, Chia-Chun Hung, Michael O' Neill, David Reiman, Kathryn Tunyasuvunakool, Zachary Wu, Akvilė Žemgulytė, Eirini Arvaniti, Charles Beattie, Ottavia Bertolli, Alex Bridgland, Alexey Cherepanov, Miles Congreve, Alexander I. Cowen-Rivers, Andrew Cowie, Michael Figurnov, Fabian B. Fuchs, Hannah Gladman, Rishub Jain, Yousuf A. Khan, Caroline MR Low, Kuba Perlin , Anna Potapenko, Pascal Savy, Sukhdeep Singh, Adrian Stecula, Ashok Thillaisundaram, Catherine Tong, Sergei Yakneen, Ellen D. Zhong, Michal Zielinski, Augustin Žídek, Victor Bapst, Pushmeet Kohli, Max Jaderberg, Demis Hassabis y John M. Jumper
- Palabra clave: Arquitectura basada en difusión, Modelado de estructuras de proteínas, Modelado del espacio biomolecular
Una función energética centrada en la columna vertebral de las redes neuronales para el diseño de proteínas
- B Huang, Y Xu, X Hu, Y Liu, S Liao, J Zhang, C Huang
- Palabra clave: función de energía, simulación MD, centrada en la columna vertebral
Diseño de proteínas de novo mediante alucinación de red profunda
- Ivan Anishchenko, Samuel J. Pellock, Tamuka M. Chidyausiku, Theresa A. Ramelot, Sergey Ovchinnikov, Jingzhou Hao, Khushboo Bafna, Christoffer Norn, Alex Kang, Asim K. Bera, Frank DiMaio, Lauren Carter, Cameron M. Chow, Gaetano T. Montelione y David Baker
- Palabras clave: alucinación, pintura interna, diseño de proteínas.
Diseño de proteínas de unión a proteínas a partir de la estructura objetivo únicamente.
- Longxing Cao, Brian Coventry, Inna Goreshnik, Buwei Huang, William Sheffler, Joon Sung Park, Kevin M. Jude, Iva Marković, Rameshwar U. Kadam, Koen HG Verschueren, Kenneth Verstraete, Scott Thomas Russell Walsh, Nathaniel Bennett, Ashish Phal, Aerin Yang, Lisa Kozodoy, Michelle DeWitt, Lora Picton, Lauren Miller, Eva-Maria Strauch, Nicholas D. DeBouver, Allison Pires, Asim K. Bera, Samer Halabiya, Bradley Hammerson, Wei Yang, Steffen Bernard, Lance Stewart, Ian A. Wilson, Hannele Ruohola-Baker, Joseph Schlessinger, Sangwon Lee, Savvas N. Savvides, K. Christopher García y David Baker
- Palabras clave: cita vinculante
Ingeniería Biomédica de la Naturaleza
- Descubrimiento antimicrobiano acelerado mediante modelos generativos profundos y simulaciones de dinámica molecular
- Payel Das, Tom Sercu, Kahini Wadhawan, Inkit Padhi, Sebastian Gehrmann, Flaviu Cipcigan, Vijil Chenthamarakshan, Hendrik Strobelt, Cicero dos Santos, Pin-Yu Chen, Yi Yan Yang, Jeremy PK Tan, James Hedrick, Jason Crain y Aleksandra Mojsilovic
- Palabras clave: antimicrobianos, autocodificador generativo, dinámica molecular.
Comunicaciones de la naturaleza
Descubriendo sustratos peptídicos de novo para enzimas mediante aprendizaje automático
- Lorillee Tallorin, JiaLei Wang, Woojoo E. Kim, Swagat Sahu, Nicolas M. Kosa, Pu Yang, Matthew Thompson, Michael K. Gilson, Peter I. Frazier, Michael D. Burkart y Nathan C. Gianneschi
- Palabras clave: diseño de enzimas, aprendizaje automático.
ECNet es un marco de aprendizaje profundo integrado en el contexto evolutivo para la ingeniería de proteínas
- Yunan Luo, Guangde Jiang, Tianhao Yu, Yang Liu, Lam Vo, Hantian Ding, Yufeng Su, Wesley Wei Qian, Huimin Zhao y Jian Peng
- Palabras clave: aptitud funcional, evolutiva.
Diseño de proteínas y predicción de variantes mediante modelos generativos autorregresivos.
- Jung-Eun Shin, Adam J. Riesselman, Aaron W. Kollasch, Conor McMahon, Elana Simon, Chris Sander, Aashish Manglik, Andrew C. Kruse y Debora S. Marks
- Palabras clave: modelos generativos autorregresivos, diseño de proteínas.
Diseño de secuencia de proteínas con potencial aprendido.
- Namrata Anand, Raphael Eguchi, Irimpan I. Mathews, Carla P. Perez, Alexander Derry, Russ B. Altman y Po-Ssu Huang
- Palabras clave: diseño de proteínas, función energética, red neuronal profunda.
Diseño de proteínas y predicción de variantes mediante modelos generativos autorregresivos.
- Jung-Eun Shin, Adam J. Riesselman, Aaron W. Kollasch, Conor McMahon, Elana Simon, Chris Sander, Aashish Manglik, Andrew C. Kruse y Debora S. Marks
- Palabras clave: modelos generativos autorregresivos, diseño de proteínas.
Inteligencia de la máquina de la naturaleza
Ciencia
ICML, ICLR o NeurIPS
Aprendizaje por refuerzo profundo para modelar complejos proteicos
- Ziqi Gao, Tao Feng, Jiaxuan You, Chenyi Zi, Yan Zhou, Chen Zhang, Jia Li
- Palabras clave: predicción de estructuras de complejos proteicos, predicción de rutas de acoplamiento, red de políticas, aprendizaje por refuerzo
BERTology se encuentra con la biología: interpretación de la atención en modelos de lenguaje de proteínas
- Jesse Vig, Ali Madani, Lav R. Varshney, Caiming Xiong, Richard Socher, Nazneen Fatema Rajani
- Palabras clave: modelo de lenguaje, transformador, propiedad estructural y funcional.
Diseño de anticuerpos condicionales como traducción de gráficos equivalentes en 3D
- Xiangzhe Kong, Wenbing Huang, Yang Liu
- Palabras clave: diseño de anticuerpos, traducción de gráficos.
Acondicionamiento por muestreo adaptativo para un diseño robusto
- David Brookes, Hahnbeom Park, Jennifer Listgarten
- Palabras clave: muestreo adaptativo, diseño de proteínas.
Los modelos generativos profundos crean estructuras proteicas nuevas y diversas
- Zeming Lin, Tom Sercu, Yann LeCun
- Palabras clave: diversidad, modelo generativo, diseño de proteínas.
Mejora profunda de las características topológicas para el diseño de proteínas de novo
- Zander Harteveld, Joshua Southern, Michaël Defferrard, Andreas Loukas, Pierre Vandergheynst, Micheal Bronstein, Bruno Correia
- Palabras clave: codificador automático variacional, características topológicas, nitidez
Fold2Seq: un modelo generativo basado en incrustación de secuencia conjunta (1D) -Fold (3D) para el diseño de proteínas
- Yue Cao, Payel Das, Vijil Chenthamarakshan, Pin-Yu Chen, Igor Melnyk, Yang Shen
- Palabras clave: modelo generativo, diseño de proteínas.
Modelado generativo de estructuras proteicas.
- Namrata Anand y Possu Huang
- Palabras clave: modelo generativo, diseño de proteínas.
Modelos generativos para el diseño de proteínas basado en gráficos
- John Ingraham, Vikas Garg, Regina Barzilay, Tommi Jaakkola
- Palabras clave: modelo generativo, diseño de proteínas.
Aprendizaje por refuerzo basado en modelos para el diseño de secuencias biológicas.
- XChristof Angermueller, David Dohan, David Belanger, Ramya Deshpande, Kevin Murphy, Lucy Colwell
- Palabras clave: aprendizaje por refuerzo, diseño de secuencia.
Generación de moléculas para la unión de proteínas objetivo con motivos estructurales
- Zaixi Zhang, Yaosen Min, Shuxin Zheng, Qi Liu
- Palabras clave: proteína diana, motivos estructurales, generación fragmento a fragmento
Aprendizaje de la representación de proteínas mediante preentrenamiento de estructuras geométricas
- Zuobai Zhang, Minghao Xu, Arian Jamasb, Vijil Chenthamarakshan, Aurelie Lozano, Payel Das, Jian Tang
- Palabras clave: descubrimiento de fármacos, diseño de fármacos, modelos generativos de nuevas estructuras moleculares.
Arxiv o bioRxiv
Generación de proteínas con difusión evolutiva: la secuencia es todo lo que necesitas
- Sarah Alamdari, Nitya Thakkar, Rianne van den Berg, Alex Xijie Lu, Nicolo Fusi, Ava Pardis Amini, Kevin K Yang
- Palabras clave: modelo de difusión, modelo generativo profundo, generación de proteínas, marco, diseño de secuencia
Un lenguaje de programación de alto nivel para el diseño generativo de proteínas.
- Brian Hie, Salvatore Candido, Zeming Lin, Ori Kabeli, Roshan Rao, Nikita Smetanin, Tom Sercu, Alexander Rives
- Palabras clave: ESMFold, modelo de lenguaje, basado en energía.
Diseño de proteínas preciso y de amplia aplicación mediante la integración de redes de predicción de estructuras y modelos generativos de difusión.
- Joseph L. Watson, David Juergens, Nathaniel R. Bennett, Brian L. Trippe, Jason Yim, Helen E. Eisenach, Woody Ahern, Andrew J. Borst, Robert J. Ragotte, Lukas F. Milles, Basile IM Wicky, Nikita Hanikel , Samuel J. Pellock, Alexis Courbet, William Sheffler, Jue Wang, Preetham Venkatesh, Isaac Sappington, Susana Vázquez Torres, Anna Lauko, Valentin De Bortoli, Emile Mathieu, Regina Barzilay, Tommi S. Jaakkola, Frank DiMaio, Minkyung Baek, David Baker
- Palabras clave: difusión, marco general de aprendizaje profundo, diseño de carpetas de novo
Diseño de proteínas guiado por funciones mediante muestreo múltiple profundo
- Vladimir Gligorijević, Daniel Berenberg, Stephen Ra, Simon Kelow, Kyunghyun Cho
- Palabras clave: codificador automático con eliminación de ruido de secuencia, muestreo múltiple profundo
Los modelos de lenguaje se generalizan más allá de las proteínas naturales.
- Robert Verkuil, Ori Kabeli, Yilun Du, Basile IM Wicky, Lukas F. Milles, Justas Dauparas, David Baker, Sergey Ovchinnikov, Tom Sercu, Alexander Rives
- Palabras clave: ESMFolk, modelo de lenguaje, diseño de red troncal fija
Los modelos lingüísticos de secuencias de proteínas a escala de evolución permiten una predicción estructural precisa
- Zeming Lin, Halil Akin, Roshan Rao, Brian Hie, Zhongkai Zhu, Wenting Lu, Allan dos Santos Costa, Maryam Fazel-Zarandi, Tom Sercu, Sal Candido, Alexander Rives
- Palabras clave: predicción de estructuras, modelo de lenguaje.
TERMinator: un marco neuronal para el diseño de proteínas basado en estructuras utilizando motivos repetidos terciarios
- Alex J. Li, Vikram Sundar, Gevorg Grigoryan, Amy E. Keating
- Palabras clave: diseño de proteínas, motivos terciarios.
Los modelos de lenguaje se generalizan más allá de las proteínas naturales.
- Robert Verkuil, Ori Kabeli, Yilun Du, Basile IM Wicky, Lukas F. Milles, Justas Dauparas, David Baker, Sergey Ovchinnikov, Tom Sercu, Alexander Rives
- Palabras clave: modelo de lenguaje, diseño de proteínas.
Otros
Referencia
[1] Wang, Jue, et al. "Andamiaje de sitios funcionales de proteínas mediante aprendizaje profundo". Ciencia 377.6604 (2022): 387-394.
[2] Huang, Bin, et al. "Una función energética centrada en la columna vertebral de las redes neuronales para el diseño de proteínas". Naturaleza 602.7897 (2022): 523-528.
Contribuyendo
Nuestro propósito es hacer que este repositorio sea aún mejor. Si está interesado en contribuir, consulte AQUÍ para obtener instrucciones sobre cómo contribuir.
Licencia
Un impresionante diseño de proteínas basado en IA se lanza bajo la licencia Apache 2.0.

