DiffAbXL
1.0.0
DiffAbXL 的實作在論文中進行了基準測試:Exploring Log-Likelihood Scores for Ranking Antibody Sequence Designs。
| 秩 | 型號 | HER2 脫落細胞 | 自然 | AZ目標2 | 大街? | ||
|---|---|---|---|---|---|---|---|
| 零射擊 | SPR控制 | 赫爾 | HER2 | ||||
| 1 | DiffAbXL-A-DN | 0.43 | 0.22 | 0.62 | 0.37 | 0.41 | 0.41 |
| 2 | DiffAbXL-A-SG | 0.46 | 0.22 | 0.64 | -0.38 | 0.43 | 0.274 |
| 3 | DiffAbXL-H3-DN | 0.49 | 0 | 0.52 | -0.08 | 0.37 | 0.26 |
| 4 | IgBlend(僅結構體) | 0.40 | 0.21 | 0.54 | -0.30 | 0.31 | 0.232 |
| 5 | 反褶皺 | 0.43 | 0.22 | 0.4 | -0.47 | 0.38 | 0.192 |
| 6 | DiffAbXL-H3-SG | 0.48 | 0 | 0.4 | -0.41 | 0.29 | 0.152 |
| 7 | 環境管理署 | 0.29 | 0 | 0 | 0.18 | 0.27 | 0.148 |
| 8 | 差異抗體 | 0.34 | 0.21 | 0 | -0.14 | 0.22 | 0.126 |
| 9 | 抗體語言2 | 0.3 | 0 | 0 | -0.07 | 0.36 | 0.118 |
| 10 | IgBlend(僅限序列) | 0.27 | 0 | 0 | -0.1 | 0.36 | 0.106 |
| 11 | 阿布朗 | 0.3 | 0 | 0 | -0.13 | 0.35 | 0.104 |
| 12 | 迪均值 | 0.37 | 0.15 | 0 | 0 | 0 | 0.104 |
| 13 | 抗體X | 0.28 | 0.19 | 0 | 0 | 0 | 0.094 |
| 14 | 抗伯特 | 0.26 | 0 | 0 | -0.17 | 0.35 | 0.088 |
| 15 | 意思是 | 0.36 | 0 | 0 | 0.02 | 0 | 0.076 |
| 16 | ESM-IF | 0 | -0.27 | 0 | -0.53 | 0.42 | -0.076 |
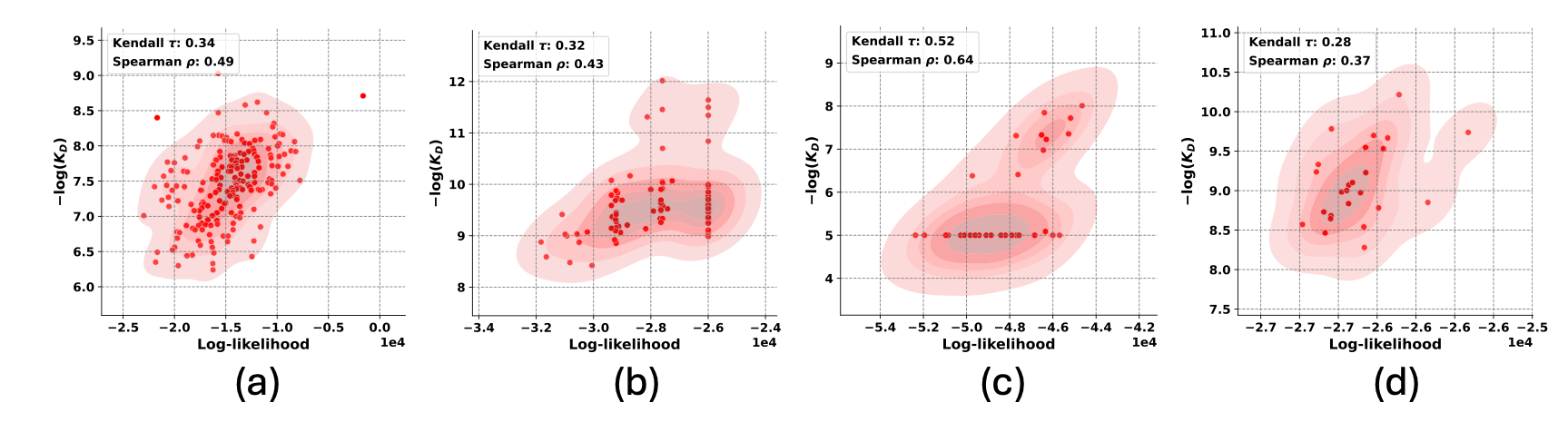
圖 1:DiffAbXL 結果: a) Absci 零樣本 HER2 資料的 DiffAbXL-H3-DN b) AZ Target-2 的 DiffAbXL-A-SG, c) Nature HEL 的 DiffAbXL-A-SG, d) DiffAbXL- Nature HER2 的A-DN。
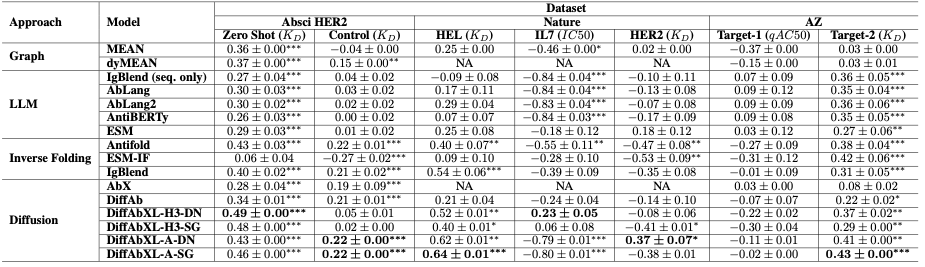
表 1: Spearman 相關性結果摘要。縮寫:DN:De Novo 模式,SG:結構指導模式,NA:需要表位或複雜結構,但不可用。 *、**、*** 分別表示 p 值低於 0.05、0.01 和 1e-4。
為了使我們更容易對您的模型進行基準測試,我們建議您在類別中將介面實作為 Python 方法,以便我們可以輕鬆地與我們的評估管道整合。此方法應接受以下輸入:
此方法應傳回一個包含以下內容的字典:
以下是用於實作此介面的 Python 基本範本:
def benchmark ( self , sequences , structure = None , mask = None , ** kwargs ):
"""
Benchmark the model on provided antibody sequences and structures.
Parameters:
sequences (list of str): List of antibody sequences.
structure (optional): Path to a PDB file. Currently, only one PDB file is provided per target dataset.
The PDB file may contain either just the antibody or an antibody-antigen complex,
depending on the dataset.
mask (optional): Binary list or array indicating the regions of interest in the sequences for metric calculations.
kwargs (optional): Additional parameters required by the model.
Returns:
dict: A dictionary containing log-likelihood scores and other relevant metrics.
"""
pass請確保您的模型以我們可以直接用於基準抗體序列設計的格式輸出對數似然分數。這將幫助我們有效地比較您的模型在我們的資料集中的表現。
有一個設定檔:sabdab.yaml,可用於變更任何參數。您可以使用以下方法訓練模型:
python train.py # For training.
- 火車.py
- 原始碼
|-model.py
- 配置
|-sabdab.yaml
- 實用程式
|-load_data.py
|-arguments.py
|-model_utils.py
|-loss_functions.py
……
- 數據
|-her2
……
權重和偏差可用於追蹤實驗。預設情況下它是關閉的,但可以透過更改./config/sabdab.yaml中設定檔中的選項來開啟
@article {Ucar2024.10.07.617023,
author = {Ucar, Talip and Malherbe, Cedric and Gonzalez Hernandez, Ferran},
title = {Exploring Log-Likelihood Scores for Ranking Antibody Sequence Designs},
elocation-id = {2024.10.07.617023},
year = {2024},
doi = {10.1101/2024.10.07.617023},
publisher = {Cold Spring Harbor Laboratory},
URL = {https://www.biorxiv.org/content/early/2024/10/24/2024.10.07.617023},
eprint = {https://www.biorxiv.org/content/early/2024/10/24/2024.10.07.617023.full.pdf},
journal = {bioRxiv}
}
如果您在自己的研究和工作中使用 DiffAbXL,請使用以下內容引用它:
@Misc{talip_ucar_2024_DiffAbXL,
author = {Talip Ucar},
title = {Exploring Log-Likelihood Scores for Ranking Antibody Sequence Designs},
URL = {https://github.com/AstraZeneca/DiffAbXL},
month = {October},
year = {since 2024}
}